核磁共振波谱学

核磁共振波谱学(英语:Nuclear Magnetic Resonance spectroscopy, NMR),简称核磁共振或核磁,是一门将核磁共振现象应用于测定物质微观结构的分析技术与学说。物质是由原子构成的,量子力学研究发现,某些原子的原子核同时具有核磁矩与核自旋带来的角动量,因此在强静态磁场下与射频电磁波会发生核磁共振现象,并产生能反映其内部结构的射频电磁波谱反馈,即核磁共振波谱。[1]:5-38 研究者对所得的核磁共振波谱进行分析,需要时可以调整样品制备、选择或设计特定的射频脉冲序列以获得特定的信息。
历经半个多世纪的发展,随着物理学界对核自旋动力学的理解逐渐深入,核磁共振波谱学产生出了众多分支技术与学说,包括最初的氢谱与碳谱,以及后续逐步发展出来的多核核磁、固态核磁[2]、二维核磁、动态核磁、同位素标记核磁等,同时也已从最初的测定分子结构[3]拓展到更广泛的各学科领域,包括材料学、晶体学[4]、生物化学[5]、分子生物学[6]、分子动力学[7]、高分子科学[8]、药剂学[9]、环境科学[10]等。
历史
早期
1920年代,物理学界通过施特恩-格拉赫实验等诸多实验认识到了电子自旋的存在,人们猜测,若原子核也存在自旋性质,原子光谱里的诸多现象就能得到解释。1939年,拉比等人将氢分子束置于静态磁场下,并给予射频电磁波辐射,发现某个特定频率的电磁波被集中吸收了,这是人类史上首次探测到核磁共振现象。1946年,斯坦福大学的布洛赫等人对核磁矩在核磁共振中的行为进行了推测,因此在样品水旁放置了能探测射频辐射的铜线圈,成功的检测到了射频能量的吸收;[11] 几乎在同一时间,哈佛大学的珀塞尔等人也用相似的方法从一小块样品蜡中探测到了氢原子磁矩对于射频辐射的吸收;[12] 这两项实验标志着核磁共振开始从理论发展为一门技术。
1948年,布隆伯根等人发现了固态核磁与液态核磁的差异,即前者的信号频率更宽,同时诠释了核弛豫概念。[13] 1949至1950年,奈特等科学家分别通过对诸如氟-19与磷-31等其他核素的核磁实验发现了化学位移的存在。[14] 1951年,帕卡德等人首次对乙醇进行了核磁实验并得到了其氢谱,这一研究向学界揭示了核磁共振能成为分析化学领域内一个强有力的技术。[15] 1952年,由于波谱仪分辨率的提高,人们发现了自旋-自旋耦合、标量耦合、化学交换等现象。[16] 同年,瓦里安联合公司推出了首个商业化的核磁共振波谱仪。[17]
初步应用
1950年代中叶,核磁共振波谱学开始在有机化学领域内得到应用,关于类固醇、糖、生物碱、卟啉等有机化合物的核磁研究相继发表,也推动了关于糖类分子手性的研究。[18] 同时,物理学界对于自旋相互作用的研究也在继续,并得到了电脑程序的辅助。1961年,瓦里安公司的“Varian A-60”波谱仪问世,大幅提高了仪器的实用性并降低了成本。新一代的波谱仪采用了双共振结构,允许对样品同时施加两个不同频率的射频场,一个可用于信号探测,另一个用于干扰样品的核自旋系统,从而允许了去耦合、核NOE效应等改进波谱分辨率的技术的运用。得益于此,碳-13核磁共振波谱学也从理论走向了实践。[19]
1950年代末,海归物理学家王义遒、丁渝、沈学础等人将核磁共振波谱学带入中国大陆,“波谱”这一译名是丁渝提出的。[20]
1966年,瓦里安公司推出了“Varian HR-220”核磁共振波谱仪,是为首个应用低温超导磁体的商业化波谱仪,大幅提高了核磁仪器的分辨率。此后,牛津大学的理查兹与牛津仪器以及布鲁克公司合作提高氢谱频率至270MHz。[21] 同年,恩斯特等人在核磁实验中运用射频脉冲,并推广了与傅里叶转换技术,大幅提高了核磁实验的灵敏度与效率。[22] 这两项技术的出现,推动了复杂有机化合物的核磁研究,例如烷烃、多肽、低聚核苷酸等。同时,固态核磁的发展也有些许突破,约翰·沃与派因斯等人将新技术成功推广到了固态领域。1971年,二维核磁的构想由吉纳提出,并由恩斯特在1970年代中叶将其发展为一项实用技术。[19]
基本原理
核磁共振
|
主条目:核磁共振 |
核自旋
一个旋转的物体具有角动量。在量子力学领域,自旋也是角动量的一种形式,然而它事实上并非由旋转产生,而是粒子的一种内禀性质,并且是量子化的。原子核由中子与质子构成,两者都具有 1/2 的自旋量子数,它们以各种数量组合构成了不同的核素。许多核素也具有自旋,核自旋量子数是由一个或多个质子与中子的自旋量子数 依照角动量叠加原理得到的,通常用 表示。同时,多个质子与中子以不同的方式叠加会产生不同的核自旋状态,核自旋态之间的跃迁通常需要极高的能量(约1011 kJ/mol),因此大多数情况下核自旋处于基态,基态核自旋量子数具有下列规律:
- 质量数为奇数的核素具有半整数自旋,例如1H,13C;
- 质量数为偶数、且质子数与中子数均为偶数的核素,基态核自旋量子数为 0,例如12C、16O、56Fe;
- 质量数为偶数、且质子数和中子数均为奇数的核素,基态核自旋量子数为大于 0 的整数,例如2H、10B、14N。
一个具有量子数为 的核量子态具有 个折叠简并态,施加磁场时,简并度会被破坏并分裂,这就是原子核的塞曼分裂。因此,诸如12C、16O等量子数为 0 的核素并不具有核磁共振行为。核自旋量子数为 1/2 的核素在核磁共振波谱学中最为重要。原子核自旋大于 1/2 的被称为四极核,由于其塞曼分裂为两个以上的能级,核磁共振研究会更加困难。[1]:5-15
| 核素 | 基态自旋 量子数 |
丰度 (%) |
磁旋比 (106 rad s-1 T-1) |
|---|---|---|---|
| 1H | 1/2 | ~100 | 267.513 |
| 2H (D) | 1 | 0.015 | 41.065 |
| 7Li | 3/2 | 92.5 | 103.962 |
| 11B | 3/2 | 80.1 | 85.847 |
| 13C | 1/2 | 1.1 | 67.283 |
| 15N | 1/2 | 0.37 | -27.126 |
| 17O | 5/2 | 0.04 | -36.281 |
| 19F | 1/2 | ~100 | 251.815 |
| 23Na | 3/2 | ~100 | 70.808 |
| 29Si | 1/2 | 4.7 | −53.190 |
| 31P | 1/2 | ~100 | 108.394 |
| 35Cl | 3/2 | 75.77 | 10.610 |
| 63Cu | 3/2 | 69.17 | 71.118 |
| 12C | 0 | 98.9 | |
| 16O | 0 | ~100 |
核的磁性
磁性是物质与磁场发生作用的能力,通常由磁矩 定量描述,一个物体的磁能取决于自身的磁矩以及磁场的大小,即 。与自旋类似,各基本粒子也具有磁性的内禀性质,原子核的磁性则是由质子与中子的磁性经量子叠加得到的,其磁矩大小与自旋角动量成正比:,其中 为磁旋比,单位为弧度每秒每特斯拉(rad s-1 T-1),是不同的粒子所具有的特征常数。[1]:23-26
进动与拉莫频率
当带有磁性的物体(例如一个指南针)被放置在磁场中时,磁矩的方向会旋转至与磁场方向平行,以降低磁能。当原子核位于磁场中时,由于其同时具有角动量,角动量与磁场的方向(即极化方向)并不能与磁场方向保持平行,而是与磁场方向保持一定角度旋转,如同在一个以磁场方向为轴的圆锥体上,这种运动方式被称为自旋进动。
自旋进动的频率被称为拉莫频率,与磁场大小和磁旋比成正比:,单位为弧度每秒(rad/s)。若以赫兹(Hz)为单位,公式如下:
进动的方向与磁旋比相关,若磁旋比为正,拉莫频率为负,进动方向则以顺时针方向沿磁场方向旋转,例如1H与13C,反之则为逆时针。[1]:26-30
共振电波
当施加射频电波(通常为脉冲波)时,进动中的核自旋会感受到两个磁场:静态磁场和射频电波的振荡场,前者的磁场强度会比后者大好几个数量级,因此通常情况下射频振荡场对核自旋状态(如磁矩的方向与大小)并不会产生影响。然而,若施加的电波频率与拉莫频率相同,两者就会发生共鸣,从而使得自旋状态发生显著变化。[1]:247 这种论述通常被称为核磁共振现象的经典物理表述。[24]
弛豫
|
主条目:弛缓 (核磁共振) |
在核自旋状态的变化过程中,弛豫时间是的一个重要因素,对其运用能很大程度上反映原子或分子的微观运动状态。
核磁实验
样品处理

核磁共振波谱仪通常由一个旋转的样品架,一个非常强的磁铁,一个射频发射器和一个接收器组成,探头(天线组件)在磁铁内部环绕样品,可选择用于扩散测量的梯度线圈和电子设备来控制系统。旋转样品是平均扩散运动所必需的。而扩散常数(扩散有序光谱法或DOSY)[25][26]的测量是在样品静止和离心的情况下进行的,流动池可用于在线分析工艺流程。
氘代NMR溶剂
NMR溶液中的绝大多数原子核属于溶剂,大多数常规溶剂是烃,并含有NMR响应的质子。 因此,氘(氢-2)被取代(99+%)。虽然氘氧化物 (D2O)和氘代DMSO(DMSO-d6)用于亲水分析物,氘代苯也是常见的,但氘代溶剂最常用的是氘代氯仿(CDCl3)。 取决于电子溶剂化效应,不同溶剂中的化学位移稍有不同。NMR波谱通常相对于已知的溶剂残余质子峰值,而不是添加的四甲基硅烷进行校准。
匀场与锁定
为了检测微弱的核磁共振射频脉冲信号,整个样品所处的静态磁场必须是十分均匀的。高分辨率核磁共振波谱仪会使用匀场片,将磁场的均匀性在几立方厘米的体积内调整至十亿分比。为了检测和补偿磁场中的不均匀性和扰动,波谱仪用一个单独的锁定单元保持对溶剂中氘频率的“锁定”,该单元本质上是一个额外的发射器和射频处理器,调谐到锁定核(氘)而不是样品的原子核。[27] 在现代核磁共振波谱仪中,均场是自动化的,尽管在某些情况下,操作员必须手动优化匀场参数以获得最佳分辨率。[28][29]
波谱分析
化学位移

|
主条目:化学位移 |
在一个分子中,各个原子的化学环境有所不同,或多或少的受到周边原子或原子团的屏蔽效应的影响,因此它们的共振频率也不同,从而导致在核磁共振波谱上,各个质子的吸收峰出现在不同的位置上。但这种差异并不大,难以精确测量其绝对值,因此人们将化学位移设成一个无量纲的相对值,即:某一物质吸收峰的频率与标准质子吸收峰频率之间的差异称为该物质的化学位移,常用 表示,单位为 ppm(百万分比)[30]。而在实际应用中,四甲基硅烷常被作为参照物,计算方法如下:
透过不同质子的化学位移,人们可以得出这些质子所处的化学环境,从而得出该分子的结构信息,这种过程称之为“解谱”。比如对于乙醇分子,具有三种不同化学环境的质子,即:甲基、亚甲基和羟基。在其1H谱图上,可以看到3个特有的峰信号各自处于特定的化学位移,其中位于1 ppm的峰信号对应甲基,位于4 ppm的信号对应亚甲基,位于2~3 ppm之间的信号对应羟基,其具体化学位移值和采用的NMR溶剂有关。另外,从峰信号的强度可以得出相对应的质子数量,比如乙醇分子中的甲基拥有3个质子,亚甲基拥有2个质子,在谱图上,对应的甲基和亚甲基峰强度比为3:2。
现代的分析软件可以协助人们通过分析峰信号,从而得出究竟有几个质子形成了此信号。这种方法称作“积分”,即通过计算面积(不单单是高度,还有峰宽度)来得出相关质子数目。但必须指出的是,这种计算方法仅适用于最简单的一维谱,对于更复杂的谱图,比如13C谱,其积分还与原子核的弛豫速率和偶极耦合常数相关,而这些常常被人误解。因此,用积分法来解析复杂核磁谱图是相当困难的。
J-耦合
| 峰的裂分 | 强度比 |
|---|---|
| 单重 | 1 |
| 双重 | 1:1 |
| 三重 | 1:2:1 |
| 四重 | 1:3:3:1 |
| 五重 | 1:4:6:4:1 |
| 六重 | 1:5:10:10:5:1 |
| 七重 | 1:6:15:20:15:6:1 |
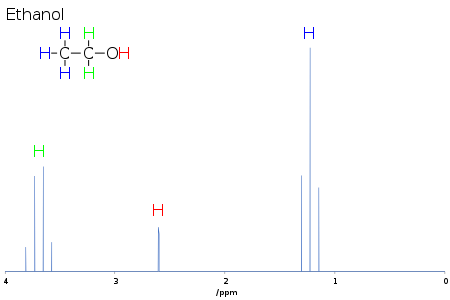
在一维谱图上,除峰信号数量,峰信号强度之外,还有一个有助于解析分子结构的信息,即磁性原子核之间的J-耦合。这种耦合来源于临近磁性原子核的不同自旋状态数的相互作用,这种相互作用会改变原子核自旋在外磁场中进动的能级分布状况,造成能级的裂分,进而造成NMR谱图中的信号峰形状发生劈裂,信号峰的劈裂状态可以得出分子内各原子和官能团之间的连接方式,以及临近的磁性核数目。
两个相邻的氢核之间的耦合遵循一定的规则,n个氢核将把相邻磁性核信号峰劈裂成n+1个多重峰,并且这n+1个多重峰之间的强度关系依照巴斯卡三角形规则。例如,乙醇分子中的甲基峰与相邻的亚甲基耦合,呈三重峰状,三重峰之间的强度比为1:2:1。不过如果一个氢核同时与两个不同性质的氢核进行耦合,则不会得到三重峰,而是得到双双重峰(dd)。要注意的是,如果两个磁性核之间相隔3个化学键以上,耦合就变得十分微弱,以至于不会出现峰的劈裂,但在芳烃和脂环类化合物中三键距离以上的长程耦合通常可以得到较复杂的劈裂峰。
19F与31P通常由于其拥有更大的自旋量子数而显得不同,其余类似,比如氢核与氘核(2D)之间的耦合将把信号峰劈裂为1:1:1。
二级耦合
上文提到的内容是在耦合常数和化学位移间隔相比很小的情况下描述的,如果耦合常数较高,或化学位移间隔小,那么多重峰的情况将变得复杂,特别是两个以上的磁性核进行耦合时,这可以通过增强多重峰当中的特定几个峰,并以牺牲其他峰的代价进行解决,不过在高场谱(比如高频谱)当中,这种现象并不明显,因此提高核磁仪器的频率可以避免此问题。
其中一些模式可以用约翰·波普(John Pople)发表的方法进行分析[31] ,尽管它的范围有限。
随着多重谱之间的频率差增加,二阶效应减小,使得高场谱(即高频谱)NMR频谱比较低频谱显示更小的失真。在早期的60 MHz频谱比通常在200 MHz或更高频率下工作的后期机器的频谱更容易失真。
磁不等价
在芳烃化合物和非弹性分子(比如烯烃)中,常会遇到由于各质子的磁不等价性而带来谱图复杂性增加,这需要计算化模型来辅助分析。
二维核磁
相关谱
|
主条目:关联性磁振频谱 |
相关谱(Correlation Spectroscopy)是二维核磁波谱的一种,常常简写为COSY。其它二维谱还包括J频谱(J-spectroscopy),交换频谱(EXSY,Exchange spectroscopy),核奥伯豪泽尔效应频谱(NOESY,Nuclear Overhauser effect spectroscopy),全相关谱(TOCSY,total correlation spectroscopy),近程碳氢相关(HSQC,Heteronuclear single quantum coherence),远程碳氢相关(HMBC,Heteronuclear multiple bond coherence)等。二维谱在解析分子结构方面可比一维谱提供更多的信息,特别是用一维谱解析复杂分子结构遇到困难的时候,二维谱可以提供帮助。历史上首次二维谱实验方法由比利时法语布鲁塞尔自由大学(Université Libre de Bruxelles)教授让·热纳(Jean Jeener)于1971年提出,之后其实验操作由沃尔特·奥厄(Walter P. Aue),恩里科·巴托尔迪(Enrico Bartholdi)和理查德·恩斯特(Richard R. Ernst)完成,并于1976年发表[32]。
固态核磁
|
主条目:固态核磁共振波谱学 |
液体核磁样品如果放在某些特定的物理环境下,是无法进行研究的,而其它原子级别的光谱技术对此也无能为力。但在固体中,像晶体,微晶粉末,胶质这样的,偶极耦合和化学位移的磁各向异性将在核自旋系统占据主导,在这种情况下如果使用传统的液态核磁技术,谱图上的峰将大大增宽,不利于研究。
目前已经有一系列的高分辨率固体核磁技术被研发出来。高解析固体核磁技术包含两个重要概念,即通过高速旋转来限制分子自取向和消除磁各向异性,对于后者,最常用的旋转方式是魔角旋转(Magic angle spinning),即旋转轴和主磁场的夹角为54.7°。
生物分子核磁
蛋白质
利用核磁谱研究蛋白质,已经成为结构生物学领域的一项重要技术手段。X射线单晶衍射和核磁都可获得高分辨率的蛋白质三维结构,不过核磁常局限于35kDa以下的小分子蛋白,尽管随着技术的进步,稍大的蛋白质结构也可以被核磁解析出来。另外,获得本质上非结构化(Intrinsically Unstructured)的蛋白质的高分辨率信息,通常只有核磁能够做到。
蛋白质分子量大,结构复杂,一维核磁谱常显得重叠拥挤而无法进行解析,使用二维,三维甚至四维核磁谱,并采用13C和15N标记可以简化解析过程。另外,NOESY是最重要的蛋白质结构解析方法之一,人们通过NOESY获得蛋白质分子内官能团间距,之后通过电脑模拟得到分子的三维结构。
核酸
“核酸核磁共振”是利用核磁共振光谱学获得关于多核酸如DNA或RNA的结构和动力学的信息。截至2003年,所有已知RNA结构中近一半已通过核磁共振波谱法确定[33] 。
核酸和蛋白质核磁共振波谱相似但存在差异。核酸具有较小的氢原子百分比,这是在NMR光谱学中通常观察到的原子,并且因为核酸双股螺旋是刚性的且大致线性的,所以它们不会自行折叠以产生“长程”相关性[34]。通常用核酸完成的NMR的类型是1H或质子NMR,13C NMR,15N NMR和31P NMR。 几乎总是使用二维核磁共振波谱方法,例如相关光谱学(COSY)和总相干转移光谱学(TOCSY)来检测穿透式核耦合,核欧佛豪瑟效应(Nuclear Overhauser effect)光谱法(NOESY)来检测彼此在空间上靠近的核之间的耦合[35]。
糖类
糖类核磁共振光谱解决糖类结构和构象的问题。
参考来源
- ^ 1.0 1.1 1.2 1.3 1.4 Malcolm H. Levitt. Spin Dynamics: Basics of Nuclear Magnetic Resonance 2nd ed. John Wiley & Sons. 2013. ISBN 978-0-470-51117-6.
- ^ Melinda J. Duer. Solid state NMR spectroscopy: principles and applications. John Wiley & Sons. 2008. ISBN 9780632053513.
- ^ Harald Günther. NMR Spectroscopy : Basic Principles, Concepts and Applications in Chemistry. John Wiley & Sons. 2013. ISBN 9783527674756.
- ^ Robin K. Harris; Roderick E. Wasylishen; Melinda J. Duer. NMR Crystallography. December 2012. ISBN 978-1-118-58732-4 (英语).
- ^ Teresa W.-M. Fan; Andrew N. Lane. Applications of NMR spectroscopy to systems biochemistry. Progress in nuclear magnetic resonance spectroscopy. 2016, 92: 18-53. doi:10.1016/j.pnmrs.2016.01.005.
- ^ Oleg Jardetzky; Gordon Carl Kenmure Roberts. NMR in molecular biology. Academic Press. 2013. ISBN 978-0123805805.
- ^ Arthur G Palmer III. NMR probes of molecular dynamics: overview and comparison with other techniques. Annual review of biophysics and biomolecular structure. 2001, 30 (1): 129-155. doi:10.1146/annurev.biophys.30.1.129.
- ^ Dan Campbell; Richard A. Pethrick; Jim R. White. Chapter 6. Polymer Characterization: Physical Techniques 2nd Edition. CRC press. 2017. ISBN 9781315274706.
- ^ Ulrike Holzgrabe. NMR spectroscopy in pharmaceutical analysis. Elsevier. 2017. ISBN 978-0-444-53173-5.
- ^ Myrna J. Simpson; Andre J. Simpson. NMR spectroscopy: a versatile tool for environmental research. John Wiley & Sons. 2014. ISBN 978-1-118-61647-5.
- ^ F. Bloch. Nuclear Induction. Physical review. 1946, 70 (7-8): 460. doi:10.1103/PhysRev.70.460.
- ^ E. M. Purcell; H. C. Torrey; R. V. Pound. Resonance Absorption by Nuclear Magnetic Moments in a Solid. Physical review: 37. doi:10.1103/PhysRev.69.37.
- ^ N. Bloembergen; E. M. Purcell; R. V. Pound. Relaxation Effects in Nuclear Magnetic Resonance Absorption. Physical review. 1948, 73: 679. doi:10.1103/PhysRev.73.679.
- ^ Samuel G. Levine. A Short History of the Chemical Shift. Journal of Chemical Education. 2001, 78 (1): 133. doi:10.1021/ed078p133.
- ^ J. T. Arnold; S. S. Dharmatti; M. E. Packard. Chemical Effects on Nuclear Induction Signals from Organic Compounds. The journal of chemical physics. 1951, 19: 507. doi:10.1063/1.1748264.
- ^ N. F. Ramsey; E. M. Purcell. Interactions between Nuclear Spins in Molecules. Physical review. 1952, 85: 143. doi:10.1103/PhysRev.85.143.
- ^ The NMR Spectroscopy Revolution. Chemical & Engineering News.
- ^ L. M. Jackman. Nuclear magnetic resonance spectroscopy in organic chemistry. Pergamon Press. 1959.
- ^ 19.0 19.1 Edwin D. Becker. A brief history of nuclear magnetic resonance. Analytical chemistry. 1993, 65 (6): 295-302. doi:10.1021/ac00054a716.
- ^ 王义遒. 对老沈的思念. 物理. 2015-03-16, 44 (4): 247 [2015-05-26]. doi:10.7693/wl20150406. (原始内容存档于2015-05-26).
- ^ David G Gadian. Sir Rex Edward Richards. 28 October 1922—15 July 2019. Biographical Memoirs of Fellows of the Royal Society (Royal Society). 2021, 70: 387-407. doi:10.1098/rsbm.2020.0039.
- ^ Richard R. Ernst; Weston A. Anderson. Application of Fourier transform spectroscopy to magnetic resonance. Review of Scientific Instruments. 1966, 31 (1): 93-102. doi:10.1063/1.1719961.
- ^ Mark J. Winter. The periodic table of the elements. 谢菲尔德大学 and WebElements Ltd, UK. [2024-05-18].
- ^ H.E Rorschach. A classical theory of NMR relaxation processes. Journal of Magnetic Resonance. 1969, 67 (3): 519-530. doi:10.1016/0022-2364(86)90388-4.
- ^ Johnson Jr., C. S. Diffusion ordered nuclear magnetic resonance spectroscopy: principles and applications. Progress in Nuclear Magnetic Resonance Spectroscopy. 1999, 34: 203–256. doi:10.1016/S0079-6565(99)00003-5.
- ^ Neufeld, R.; Stalke, D. Accurate Molecular Weight Determination of Small Molecules via DOSY-NMR by Using External Calibration Curves with Normalized Diffusion Coefficients. Chem. Sci. 2015, 6: 3354–3364. doi:10.1039/C5SC00670H.

- ^ Nuclear Magnetic Resonance Spectroscopy Center | Washington State University.
- ^ Center for NMR Spectroscopy: The Lock. nmr.chem.wsu.edu.
- ^ NMR Artifacts. www2.chemistry.msu.edu.
- ^ 詹姆斯·奇勒. 第二章:核磁共振与能级 (剑桥大学重印). 理解核磁共振波谱. 尔湾加州大学. [2007-05-11]. (原始内容存档 (PDF)于2020-01-09).
- ^ Pople, J.A.; Bernstein, H. J.; Schneider, W. G. The Analysis of Nuclear Magnetic Resonanace Spectra. Can J. Chem. 1957, 35: 65–81.
- ^ Martin,G.E; Zekter,A.S., Two-Dimensional NMR Methods for Establishing Molecular Connectivity; VCH Publishers,Inc: New York,1988 (p.59)
- ^ Fürtig, Boris; Richter, Christian; Wöhnert, Jens; Schwalbe, Harald. NMR Spectroscopy of RNA. ChemBioChem. 2003, 4 (10): 936–62. PMID 14523911. doi:10.1002/cbic.200300700.
- ^ Addess, Kenneth J.; Feigon, Juli. Introduction to 1H NMR Spectroscopy of DNA. Hecht, Sidney M. (编). Bioorganic Chemistry: Nucleic Acids. New York: Oxford University Press. 1996. ISBN 0-19-508467-5.
- ^ Wemmer, David. Chapter 5: Structure and Dynamics by NMR. Bloomfield, Victor A.; Crothers, Donald M.; Tinoco, Ignacio (编). Nucleic acids: Structures, Properties, and Functions. Sausalito, California: University Science Books. 2000. ISBN 0-935702-49-0.
外部链接
核磁技术资讯网站
- 蛋白质核磁实践指导 (页面存档备份,存于互联网档案馆)
- 詹姆斯·奇勒. 理解核磁共振波谱 (剑桥大学重印). 尔湾加州大学出版. [2007-05-11]. (原始内容存档于2013-06-30).
- 核磁共振波谱基础 (页面存档备份,存于互联网档案馆) - 核磁共振理论, 设备和技术总览. (RIT化学教授约瑟夫·霍尔纳克博士 著)
- 核磁维基网站 (页面存档备份,存于互联网档案馆) 基于维基的核磁项目网站
- 有机化学中的核磁共振波谱
- 中国核磁资源库
核磁处理、分析和模拟资源
- WINDNMR-Pro (页面存档备份,存于互联网档案馆) 一个模拟软件, 用于自旋耦合的一二级图谱的交互式计算
- CARA - 乌特里希工作组开发的共振工作软件
- NMRShiftDB (页面存档备份,存于互联网档案馆) - 核磁谱图预测和开放数据库网站
- Spinworks (页面存档备份,存于互联网档案馆)
- SPINUS (页面存档备份,存于互联网档案馆) 利用神经网络来预测化学分子结构核磁谱图的网站
| ||||||||||||||||||||||||||||||||
| |||||||
| ||||||||||||
Text is available under the CC BY-SA 4.0 license; additional terms may apply.
Images, videos and audio are available under their respective licenses.











