分子进化
| 演化生物学 |
|---|
 |
|
分子演化是指细胞分子(如DNA、RNA和蛋白质)的序列组成在不同世代间发生改变,或是指研究此现象的学门。此研究领域主要使用演化生物学和族群遗传学的原理来解释分子演化的规律,主要的研究主题有点突变的发生率和影响、中性漂变和自然选择的相对重要性、新基因的起源、复杂性状的可遗传性、物种形成的遗传基础、发育过程的演化、以及演化力量对基因体及性状的影响。
发展史
分子演化发源自20世纪初的“比较生物化学”,在1960至70年代成形。蛋白质定序技术的发展让分子生物学家得以用序列比对来进行种系发生的研究,并用同源序列的差异程度建立分子时钟来推估最后共同祖先的存在时间。1960年代末,中性理论的发展让分子时钟有了理论基础,并和主张自然选择是主要演化动力的学者们产生激烈争辩。1970年代,核酸定序的技术让分子演化进一步推展到高度保守的核糖体RNA,让生物学家可以推论早期的生命演化史。
早期
在分子生物学于1950~1960年代成形之前,透过比较物种间的生化差异来研究演化的可能性只有少数生物学家探究过。艾弗德·史都特方在1921年预测到染色体反转,并和费奥多西·多布然斯基在1937年首次在Drosophila Pseudo-obscura果蝇中用染色体反转的数量重建了17个品系的种系发生史[1]。厄尼斯特·鲍德温(Ernest Baldwin)的比较生物化学研究在1930年代开始;1940年代马歇·弗洛金(Marcel Florkin)首创用分子和生化特征重建种系发生的技术。但一直到1950年代生物学家才发展出能收集生化资料作为分子演化计量研究的方法[2]。
第一个分子系统分类学研究是用免疫分析和蛋白指纹技术达成。1954年艾伦·博伊登(Alan Boyden)由乔治·努特尔(George Nuttall)的免疫学技术延伸出新的技术。在1960年代初,寇地思·威廉斯(Curtis Williams)和莫里斯·古德曼(Morris Goodman)便用免疫比对来研究灵长类的种系发生。莱纳斯·鲍林和他的学生则兼用新发明的电泳和纸质层析来分析部分消化的蛋白质,而建立了二维的资讯,这让同源蛋白质的比较更加精细。[3]
1950年代也有一些自然学家尝试了分子技术,如恩斯特·迈尔和查理斯·希伯利(Charles Sibley)。迈尔放弃了纸质层析,但希伯利用电泳分析蛋白蛋白质进行鸟类分类,并辅以DNA杂合,开启了他一生在分子系统分类学的研究。[4]
虽然这些早期的研究在演化生物学界有些许摩擦,但整体而言没造成太大的影响,直到分子生物学对基因的本质提供了新的观点。
遗传负荷、古典/平衡争议、以及异型合子的量测
1950年代分子生物学开始成形时,有个争辩了很久的议题——古典/平衡争议——是关于杂种优势的成因。1950年·詹姆斯·F·克罗(James F. Crow)提供了两个可能解释,后来被称为“古典派”和“平衡派”。“古典派”立场可溯源自J·B·S·霍尔丹在1937年提出的难题:有害突变对一个族群的平均适应力的影响只取决于突变率,因为危害较重的突变都会迅速被天择移除,只有危害较轻的突变会维持较久,H·J·马勒称之为遗传负载。马勒进一步主张杂种优势是因为生物带有有害的隐性同型合子,这些有害的基因在杂交时会被显性的等位基因隐藏。依此论点,天择会偏好同型合子,放射线等引发突变的因子只会提高遗传负载。而“平衡派”主要来自布鲁斯·华勒斯(Bruce Wallace)和杰克·L·金恩(J. C. King),主张超显性(异型合子带有较高的适应度)才是杂种优势的成因。依此论点,天择会偏好异型合子,而放射线可以增加异型合子而提高族群的适应度。[5]
这个争议在1950年代渐渐成为族群遗传学的核心问题。1958年华勒斯的果蝇研究显示放射线诱发的突变提高了本来是纯品系的果蝇,为平衡派立场提供了证据。华勒斯并估算野生果蝇应有约一半的基因座是异型合子。木村资生的数学分析则显示就算超显性基因座很少,它们仍可能解释很大比例的基因多样性,因此他和他的导师克罗持古典派立场。克罗和木村资生更进一步提出无限等位基因模型(infinite alleles model),可以依据族群大小、突变率以及天择对突变的影响来推算等位基因的数量。这表示只要异型合子的比例可以量测出来,就能用来分辨古典派或平衡派何者较正确[6]。
到了1960年代中期,蛋白质电泳等生物化学和分子生物学技术允许生物学家测量自然族群的异型合子比例(heterozygosity)。量测的结果发现果蝇有很大比例的异型合子(平均为12%)[7][8]。但结果却引发不小争议,因为当时主流不认为中性的基因占多数,而结果虽支持平衡派的解释,但这解释会造成很大的分离负载(segregation load)[9]。
蛋白质序列和分子时钟
演化生物学家和分子生物学家开始朝对方的领域发展。
1951~1955年间用胰岛素建立蛋白质定序的基本技术后,弗雷德里克·桑格等人在1956年发表了一些跨物种的序列比对·弗朗西斯·克里克和查理斯·希伯利(Charles Sibley)等人看出了用生物序列重建种系发生史的可能。至1960年代初期,蛋白质定序的技术已经允许比较同源氨基酸序列。1961年伊曼纽·马戈列许(Emanuel Margoliash)等人定序了马和另外几种生物的细胞色素c。[10]
1962年莱纳斯·鲍林和艾美·祖柯坎(Emile Zuckerkandl)提出了分子时钟,透过比较同源蛋白质的差异来推算双方分歧的时间。祖柯坎将他想到的概念用在鲍林的研究的血红素上,当时正由瓦特·薛德(Walter Schroeder)定序。结果不但符合已知的脊椎动物种系发生史,还进一步支持一个1957年提出的假说:同一个生物内的不同血红素蛋白是同源的[11]。从1962至1965年间两人进一步完善了分子时钟的理论,而艾莫·P·史密斯(Emil L. Smith)和马戈列许将分析研伸到细胞色素c。这些研究和当时基于古生物学建立的已知种系发生时间吻合,但是分子时钟的基本假设——蛋白质的演化速率很一致——是非常有争议的。[12]
分子演化的机制
基因体的结构和内容是受分子生物学和族群遗传学的力量影响,新的基因由突变产生,并因遗传漂变或天择而改变它在族群中的频率,可能完全取代其他的基因,或是消失。
突变
|
主条目:突变 |
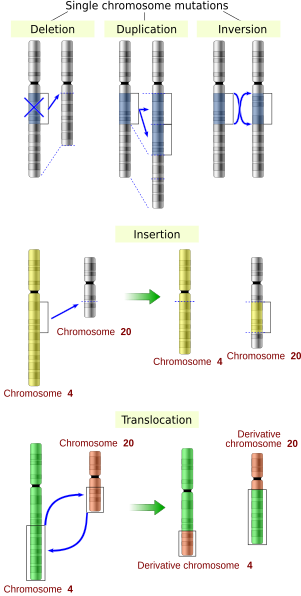
突变是细胞或病毒的遗传物质上永久(但可逆)、可遗传的改变,是唯一能产生新的基因的方式。突变源自细胞分裂时DNA复制的错误,或接触到放射线、特定致癌物、环境压力、或病毒和跳跃子。大多数突变是造成单核苷酸多态性的点突变,只改变核酸序列上的单一个碱基。另外也有较大规模的突变如复制(duplication)、插入(insertion)、删除(deletion)、反转(inversion)和转移(translocation)。[13]
各种点突变发生的几率不同,会影响GC含量。点突变可分为同类置换(transition,又译转换,指腺嘌呤和鸟嘌呤或胞嘧啶和胸腺嘧啶之间的替换)和异类置换(transversion,又译颠换,指嘌呤和嘧啶之间的替换),其中同类置换较常发生,而异类置换则较常造成蛋白质的氨基酸序列改变。[13]
点突变是随机发生的,通常几率很低。在大多数生物中,单一核苷酸的突变率很低,约在每世代每核酸10−9~10-8,不过在病毒中有时可高达10−6[14]。因为突变很少发生,它们是很缓慢地随着世代累积。随然每个世代产生的突变数目并不固定,但长时间下来突变会以固定的速率累绩。比较序列之间单核苷酸多态性的数量,除以每世代的平均累积的突变数量,可以估算出两个支系分歧了多久,此方法称为分子时钟。[15]
取决于新突变的相对适应度,新的突变可能被不同的演化力量影响。如果新突变对个体有利或有害,会受天择影响,如果是中性(不影响适应度),则主要受遗传漂变影响。参见下方“各机制的相对重要性”一节。
遗传重组
|
主条目:遗传重组 |
遗传重组会交换不同染色体或同一染色体的不同区域之间的基因,而打破遗传连锁,让基因能够自由组合,并降低遗传便车效应(genetic hitchhiking)。这使得天择更有效率,因此,重组率高的区域会保留较少有害突变、较多有利的等位基因、和较少DNA复制的错误。[16]
重组也可能造成突变。当染色体对齐失误时,重组可能造成许多种大范围的突变。另外还有一种机制称为基因转换(Gene conversion),是指DNA修复时,在切除错误的碱基后,使用另一个同源基因作为互补模板来进行DNA修复,合成修复的DNA。基因转换能造成等位基因在配子中偏离1:1的比例,除此之外,还会造成同源基因之间的差异下降,降低核酸分歧的程度,称为协同演化(concerted evolution)。[17]
遗传漂变

|
主条目:遗传漂变 |
遗传漂变是因为族群大小有限时,随机采样造成等位基因频率在世代间随机改变。即使不同等位基因有相同的适应度,它们仍会因几率而提高或降低频率,直到频率降到0(丢失)或升到1(固定)。就算适应度不同,若差异不大或是有效族群大小很小,这些近中性的变异仍会受到遗传漂变的影响。许多基因体的特征都可能漂变作用在近中性变异上的结果[18]。
选择
当生物的适应度不同时,即会发生选择,也就是说当一个基因能让个体更能存活、竞争配偶或生育后代时,这种个体会产生较多后代,因此下一代会有较多这种基因。选择包括自然选择、人工选择和性选择。自然选择源自个体对其周遭有机或无机的自然环境有不同的适应度,性选择源自择偶偏好,有些基因能帮助个体得到异性的青睐,而不见得会帮助个体适应环境。人工选择即育种是人为选殖偏好的特征。
各种选择在族群遗传学上的原理是类似的,但对分子演化的影响不尽相同,因为不同功能的基因常有不同的分布,例如性征的基因常分布在性染色体上,因此性选择也较常影响性染色体的演化。
选择可以作用在基因而非个体的层级,即使降低个体的适应度,自私DNA仍然可以演化,造成基因组内部冲突。例子包括跳跃子、减数分裂驱动者(meiotic drivers)、杀手X染色体(killer X chromosomes)、自私线粒体(selfish mitochondria)、自我复制内含子 (self-propagating introns)。这对基因体的演化有重大影响[19]。
各机制的相对重要性
基于前述各机制的相对重要性,分子演化可以分出三大派观点[20]。
天择假说主张自然选择是分子演化的主要力量。此理论同意许多突变是中性的,但认为这些基因的频率主要受其他与之有连锁不平衡的基因上的天择影响,而不是遗传漂变[21]。此假说也认为微弱的天择足以解释密码子偏差(Codon usage bias)[22] 。
中性假说强调突变、净化选择(purifying selection)和遗传漂变[23]。木村资生建立的中性理论称大多数的突变是有害的,会快速被天择移除,余下的突变中,大多数则是中性的(意即不影响适应度),只有非常少数会提高适应度。这些占多数的中性突变是受遗传漂变影响,并解释核苷酸多态性和物种差异[24][25][26]。这个理论很快地得到杰克·L·金恩(Jack L. King)和汤玛士·H·秋克斯(Thomas H. Jukes)的研究支持[27],让学界激烈争辩新达尔文主义在分子演化的重要性。此理论的一个变形是近中性理论,认为大多数突变接近中性,但不是完全中性,因此随机漂变和天择都强烈影响分子演化[25]。
突变假说则强调突变在分子机制上的特定偏向[28]。第一位提出现代突变论的是末冈登(Naboru Sueoka),他主张GC含量的差异不是天择的影响,而是GC突变压力的结果[29]。
基因体的演化
|
主条目:基因体的演化 |
基因体的各种特征,包括其大小、序列特征、染色体数目与结构、基因在基因体中的分布等等,都是分子演化的重要主题。
基因体大小
基因体的大小取决于基因的数量和重复序列的多寡。基因体的大小——又称C值——和生物的复杂程度并没有相关,这个看似不合理的现象称为C值谜[30]。这个谜题的解释有二:首先,不会转录非编码DNA如重复序列可以占非常大的比例,让基因体变得很大;再者,不是每个基因都对应到特定的发育阶段或组织类型,很多基因功能重复或、不会表现、或是只调节基他基因,也会让基因体变大而不增加生物的复杂度。[31]
包括哺乳类在内的许多真核生物都带有大量的非编码DNA,而基因体大小和大多数生理特征都没有关连。一般认为,真核生物的基因体大小受到的天择不强,因此许多突变都是近中性的,重复序列和其他非编码可以在族群中累积而影响个体的适应力。不过鸟类的基因体较小,重复序列很少,这可能是因为鸟类的红血球有细胞核,而越大的细胞核能带的氧气越少,但鸟类是内温动物而且会飞行,为了维持够高的代谢率产生足够的能量,红血球必需有效率地运送氧气,这造成鸟类的C值受到较强的天择。有间接证据显示恐龙的基因体也偏小,符合其内温调节和跑步的特征[32]。许多原核生物的基因体也受天择影响,因为较大的基因体在DNA复制较花时间,也比较耗能,影响个体的适应度。
重复序列和跳跃子

DNA序列里面可以发现许多不断重复的序列。其分布连续在一起的是局部分布的重复序列(localized repeated sequences)串联重复序列(tandem repeat),可再依单元序列的长短分为卫星序列(satellite)、小卫星序列(minisatellite)和微卫星序列(microsatellite);分布较为分散的是散布重复序列(interspersed repeat),可以细分为短散落元件(short interspersed nuclear element)和长散落元件(long interspersed nuclear element)。在灵长目中,最常见的短散落元件是Alu元件,长散落元件是CRISPR/Cas 系统。[33][34]
很多散布重复是跳跃子,即可以在基因体中自我复制并移动位置的序列;许多跳跃子序列和病毒序列类似[35]。跳跃子的演化是以基因为单位的演化,天择直接作用在跳跃子本身,而非带有跳跃子的个体。跳跃子对基因体的演化有重大影响。[19]
基因内容和分布
不同物种带有不同数量的基因,以不同的模式散布在其基因体内。有些物种,如大多数细菌、Drosophila属果蝇、拟南芥的基因体特别紧凑,非编码DNA较少。相较之下,哺乳动物和玉米的基因体则有大量的重复序列、较长的内含子以及非编码DNA。基因的内容和分布会改变其突变率,而影响该基因以及整个物种的演化。带有较长内含子会使编码序列之间的距离增加,而使基因比较容易发生重组,包括异位重组(ectopic recombination), 也因此容易造成新基因的形成。[30]
染色体数目与结构
一个生物的基因体内的染色体数目在物种之间有非常大的变化,并和其DNA长度并不相关。例如杰克跳蚁(Myrmecia pilosula)只有一对染色体[36],而心脏叶瓶尔小草有1260对染色体[37]。纤毛虫基因体内的每一个基因都有独立的一个染色体, 因此基因之间都没有遗传连锁,这让天择的效率大为提高。[38]
染色体数目的演化对物种形成可能有举足轻重的影响,因为不同的染色体数目会造成生殖隔离。人类2号染色体是融合自两条黑猩猩的染色体,仍带有位在中间的端粒以及第二个残留的着丝粒[39]。植物中常出现多倍体,特别是异源多倍体(allopolyploidy),造成杂交后代和任一亲代产生生殖隔离,于是在一个世代内形成同域种化。点灰蝶属蝴蝶的染色体数目差异极大,从10对到134对都有,该属也是种化频率最高的支系之一[40]。
胞器基因
除了细胞核内的基因,内共生胞器也有自己的基因,通常是环状的质体。线粒体和叶绿体DNA在不同类群中并不相同,但膜蛋白——尤其是和电子传递链的构成分子——通常编在胞器基因中。大多数生物的线粒体和叶绿体是母系遗传,因为这些胞器存在较大的卵中,而不是精子。在少数特例(如一些孔雀蛤)中,线粒体是父系遗传。[41]
新基因的起源
新的基因座可以由许多种不同的机制产生,包括基因复制、反转录复制转移、基因嵌合(chimeric gene formation)、基因截短(gene truncation)、以及招收非编码序列等直接形成(de novo origination)机制。[42]
基因复制发生时,一段基因序列产生另一份复制,而有了冗余重复,重复的基因可以突变出不同功能而不影响原本的功能,或是各段复制各自专精于原始功能的一部分。除了整个基因的复制,有时也会发生基中当中的一部分复制,变成更长的基因。

反转录转移将信使RNA反转录成DNA并插回基因体中。反转录基因常插入到新的位置,而有了不同的基因表现和功能。
嵌合基因(Chimeric genes)是当复制、删除或反转录转移造成两个不同的编码序列嵌合在一起,而形成新的基因序列。嵌合基因常造成调节过程的改变,并可能重新排列蛋白质域来产生新的功能。
直接形成机制已在酵母菌[43] 、稻米[44]、以及人类[45] 中发现。直接形成可以从已经低度表现的可转录序列形成[46],终止密码子突变成其他密码子或是发生框架转移突变(frameshift mutation)时,则会形成延长的蛋白质,包含原本属于非编码序列的部分。招收非编码DNA是另一个直接形成新基因的机制[47]。例如有果蝇(Drosophila melanogaster)的基因体中有至少五个新基因是如此形成[48][49] 。
分子分类学
|
主条目:分子分类学和种系发生学 |
分子分类学结合传统的系统分类学和分子遗传学,用DNA、RNA或蛋白质序列来回答系统分类学的问题,即从演化生物学的角度找出正确的生物分类法或生物分类学[50]。一般而言使用分子分类需要约1000个碱基长度的序列。分子分类学的建立是奠基在许多技术的发展上,如DNA定序可以准确得知DNA或RNA的碱基。将整个基因体定序的成本正快速下降中,因此完全定序的物种数量正呈指数上升[51]。
和形态演化的不同步
特别是在在单系胞生物中,分子演化和形态演化有时并不同步,会出现“单一形态多重支系”(例如趋同演化和隐存种)和“单一支系多重形态”(例如性状可塑性和不同生命周期)。有些案例可以用中性理论解释[52] 。
学术界
社群和期刊
分子生物学与演化社群(Society for Molecular Biology and Evolution定期连载《分子生物学与演化》(Molecular Biology and Evolution)和《基因体生物学与演化》(Genome Biology and Evolution)两份期刊,并举办年会[53]。此领域的其他期刊还有《分子演化期刊》(Journal of Molecular Evolution)和《分子分类和演化》(Molecular Phylogenetics and Evolution)。分子演化的研究也常发表在遗传学、分子生物学、基因体学、系统分类学或演化生物学的学术期刊。
知名学者
- 木村资生:提出中性理论。
- 杰克·L·金恩(Jack L. King):为中性理论提供佐证。
- 汤玛士·H·秋克斯(Thomas H. Jukes):为中性理论提供佐证。
- 太田朋子:提出近中性理论;木村资生的门生。
- 根井正利:发展中性理论;木村资生的门生。
- 李文雄:分子时钟和基因复制的研究;中央研究院院士、美国国家科学研究院院士,根井正利的门生。
- 吴仲义:物种形成的基因体研究;中央研究院院士、美国科学促进会会士,根井正利的门生。
- 约翰·李·哈比(John Lee Hubby):发明胶体电泳,在果蝇的研究为分子演化域领打下基础。
- 理查·勒翁亭(Richard Lewontin):在果蝇的研究为分子演化域领打下基础。
- 莱纳斯·鲍林:提出分子时钟,诺贝尔化学奖得主。
- 艾美·祖柯坎(Emile Zuckerkandl):提出分子时钟。
- 麦柯·林区(Michael Lynch):基因体结构的研究。
- 约瑟夫·费尔森斯坦:族群遗传学的理论研究。
- 约翰·伯顿·桑德森·霍尔丹:杂交和种化的研究。
重要著作
- Li, W.-H. Molecular Evolution. Sinauer. 2006. ISBN 0-87893-480-4.
- Michael Lynch. The Origins of Genome Architecture. Sinauer. 2007. ISBN 0-87893-484-7.
- A. Meyer (Editor), Y. van de Peer, "Genome Evolution: Gene and Genome Duplications and the Origin of Novel Gene Functions", 2003, ISBN 978-1-4020-1021-7
- T. Ryan Gregory, "The Evolution of the Genome", 2004, YSBN 978-0123014634
参见
参考资料
- ^ Dobzhanski, Sturtevant, 1937
- ^ Dietrich, "Paradox and Persuasion", pp. 90-91; Zuckerkandl, "On the Molecular Evolutionary Clock", p. 34
- ^ Dietrich, "Paradox and Persuasion", pp. 90-91; Morgan, "Emile Zuckerkandl, Linus Pauling, and the Molecular Evolutionary Clock", pp. 161-162.
- ^ Hagen, "Naturalists, Molecular Biologists, and the Challenges of Molecular Evolution", pp. 335-339
- ^ Dietrich, "The Origins of the Neutral Theory of Molecular Evolution", pp. 25-31
- ^ Dietrich, "The Origins of the Neutral Theory of Molecular Evolution", pp. 33-41
- ^ Hubby, J. L. Protein Differences in Drosophila. I. Drosophila melanogaster. Genetics. 1963, 48 (6): 871–879. PMC 1210521
 . PMID 17248176.
. PMID 17248176.
- ^ Hubby, J. L.; Lewontin, R. C. A Molecular Approach to the Study of Genic Heterozygosity in Natural Populations. I. The Number of Alleles at Different Loci in Drosophila pseudoobscura. Genetics. 1966, 54: 546–595.; and Lewontin, R. C.; Hubby, J. L. A Molecular Approach to the Study of Genic Heterozygosity in Natural Populations. II. Amount of Variation and Degree of Heterozygosity in Natural Populations of Drosophila pseudoobscura. Genetics. 1966, 54 (2): 595–609. PMC 1211186
 . PMID 5968643.
. PMID 5968643.
- ^ Dietrich, "The Origins of the Neutral Theory of Molecular Evolution", pp. 42-45
- ^ Hagen, "Naturalists, Molecular Biologists, and the Challenge of Molecular Evolution", pp. 323-325
- ^ Zuckerkandl, "On the Molecular Evolutionary Clock", pp. 34-35
- ^ Dietrich, "Paradox and Persuasion", pp. 91-94
- ^ 13.0 13.1 Bergstro, Carl T. & Dugatkin, Lee Alan. Evolution. New York: W. W. Norton & Company. 2012: 190-193. ISBN 978-0-393-92592-0.
- ^ Michael Lynch. Evolution of the mutation rate. Trends in Genetics. August 2010, 26 (8): 345-352 [2015-07-16]. doi:10.1016/j.tig.2010.05.003. (原始内容存档于2018-12-11).
- ^ Daniel L. Hartl & Andrew G. Clark. Principles of Population Genetics. Sunderland, MA, USA: Sinauer Associates. 2007: 329-340. ISBN 978-0-87893-308-2.
- ^ Douglas J. Futuyma. Evolution 2. Sinauer Associates. 2009: 193, 232-233. ISBN 978-0-87893-223-8.
- ^ Douglas J. Futuyma. Evolution 2. Sinauer Associates. 2009: 193, 547. ISBN 978-0-87893-223-8.
- ^ Michael Lynch. The Origins of Genome Architecture. Sinauer. 2007. ISBN 0-87893-484-7.
- ^ 19.0 19.1 David J. Finnegan. Eukaryotic transposable elements and genome evolution. Trends in Genetics. 1989, 5: 103-107 [2015-07-16]. ISSN 0168-9525. doi:10.1016/0168-9525(89)90039-5. (原始内容存档于2018-12-11).
- ^ Graur, D. and Li, W.-H. Fundamentals of molecular evolution. Sinauer. 2000. ISBN 0-87893-266-6.
- ^ Hahn, Matthew W. TOWARD A SELECTION THEORY OF MOLECULAR EVOLUTION. Evolution. February 2008, 62 (2): 255–265. PMID 18302709. doi:10.1111/j.1558-5646.2007.00308.x.
- ^ Hershberg, Ruth; Petrov, Dmitri A. Selection on Codon Bias. Annual Review of Genetics. December 2008, 42 (1): 287–299. PMID 18983258. doi:10.1146/annurev.genet.42.110807.091442.
- ^ Kimura, M. The Neutral Theory of Molecular Evolution. Cambridge University Press, Cambridge. 1983. ISBN 0-521-23109-4.
- ^ Nachman M. C.W. Fox and J.B. Wolf , 编. "Detecting selection at the molecular level" in: Evolutionary Genetics: concepts and case studies: 103–118. 2006.
- ^ 25.0 25.1 Ohta, T. The nearly neutral theory of molecular evolution. Annual Review of Ecology and Systematics. 1992, 23 (1): 263–286. doi:10.1146/annurev.es.23.110192.001403.
- ^ Kimura, Motoo. Evolutionary rate at the molecular level (PDF). Nature. 1968, 217 (5129): 624–626 [2015-07-16]. Bibcode:1968Natur.217..624K. PMID 5637732. doi:10.1038/217624a0. (原始内容存档 (PDF)于2016-03-03).
- ^ King, J.L. and Jukes, T.H. Non-Darwinian Evolution (PDF). Science. 1969, 164 (3881): 788–798 [2015-07-16]. Bibcode:1969Sci...164..788L. PMID 5767777. doi:10.1126/science.164.3881.788. (原始内容存档 (PDF)于2019-12-31).
- ^ Nei, M. Selectionism and Neutralism in Molecular Evolution. Molecular Biology and Evolution. 2005, 22 (12): 2318–2342. PMC 1513187
 . PMID 16120807. doi:10.1093/molbev/msi242.
. PMID 16120807. doi:10.1093/molbev/msi242.
- ^ Sueoka, N. On the evolution of informational macromolecules. In: Bryson, V. and Vogel, H.J. (编). Evolving genes and proteins. Academic Press, New-York. 1964: 479–496.
- ^ 30.0 30.1 Thomas Jr, C. A. The genetic organization of chromosomes (PDF). Annual review of genetics. 1971, 5 (1): 237-256 [2019-01-02]. (原始内容 (PDF)存档于2013-10-23).
- ^ Sean Eddy (2012) The C-value paradox, junk DNA, and ENCODE (页面存档备份,存于互联网档案馆), Curr Biol 22(21):R898–R899.
- ^ Organ, C. L.; Shedlock, A. M.; Meade, A.; Pagel, M.; Edwards, S. V. Origin of avian genome size and structure in nonavian dinosaurs. Nature. 2007, 446: 180–184. doi:10.1038/nature05621.
- ^ Medical Subject Headings. National Library of Medicine. 2011 [2015-07-15]. (原始内容存档于2016-04-07).
- ^ 郑杏倩. 微衛星DNA. 科技部高瞻自然科学教学资源平台. 2013-08-06 [2015-07-16]. (原始内容存档于2020-07-24).
- ^ Thomas Wicker, François Sabot, Aurélie Hua-Van, Jeffrey L. Bennetzen, Pierre Capy, Boulos Chalhoub, Andrew Flavell, Philippe Leroy, Michele Morgante, Olivier Panaud, Etienne Paux, Phillip SanMiguel & Alan H. Schulman. A unified classification system for eukaryotic transposable elements. Nature Reviews Genetics. December 2007, 8: 973-982 [2015-07-16]. doi:10.1038/nrg2165. (原始内容存档于2012-01-05).
- ^ Crosland, M.W.J., Crozier, R.H. Myrmecia pilosula, an ant with only one pair of chromosomes. Science. 1986, 231 (4743): 1278. Bibcode:1986Sci...231.1278C. PMID 17839565. doi:10.1126/science.231.4743.1278.
- ^ Gerardus J. H. Grubben. Vegetables. PROTA. 2004: 404 [10 March 2013]. ISBN 978-90-5782-147-9. (原始内容存档于2014-09-24).
- ^ Robert S. Coyne, Douglas L. Chalker, and and Meng-Chao Yao. GENOME DOWNSIZING DURING CILIATE DEVELOPMENT:Nuclear Division of Labor through Chromosome Restructuring. Annual Review of Genetics. December 1996, 30: 557-578 [2015-07-16]. doi:10.1146/annurev.genet.30.1.557. (原始内容存档于2020-07-24).
- ^ Ijdo, J. W., Baldini, A., Ward, D. C., Reeders, S. T., & Wells, R. A. Origin of human chromosome 2: an ancestral telomere-telomere fusion. Proceedings of the National Academy of Sciences. 1991, 88 (20): 9051-9055.
- ^ Nikolai P. Kandul, Vladimir A. Lukhtanov, Naomi E. Pierce, KARYOTYPIC DIVERSITY AND SPECIATION IN AGRODIAETUS BUTTERFLIES, The Society for the Study of Evolution, 2007,, 61(3):546-559, doi:10.1111/j.1558-5646.2007.00046.x
- ^ Sophie Breton, Hélène Doucet Beaupré, Donald T. Stewart, Walter R. Hoeh, Pierre U. Blier. The unusual system of doubly uniparental inheritance of mtDNA: isn't one enough?. Trends in Genetics. September 2007, 23 (9): 465-474, [2015-07-16]. ISSN 0168-9525. doi:10.1016/j.tig.2007.05.011. (原始内容存档于2018-12-11).
- ^ D. V. Babushok, E. M. Ostertag, H. H. Kazazian Jr. Current topics in genome evolution: Molecular mechanisms of new gene formation. Cellular and Molecular Life Sciences. 2006-12-27, 64 (5): 542-554.
- ^ Cai J; Zhao R; Jiang H; et al. De novo origination of a new protein-coding gene in Saccharomyces cerevisiae. Genetics. 2008, 179 (1): 487–496. PMC 2390625
 . PMID 18493065. doi:10.1534/genetics.107.084491.
. PMID 18493065. doi:10.1534/genetics.107.084491.
- ^ Xiao W; Liu H; Li Y; et al. El-Shemy, Hany A , 编. A rice gene of de novo origin negatively regulates pathogen- induced defense response. PLoS ONE. 2009, 4 (2): e4603. Bibcode:2009PLoSO...4.4603X. PMC 2643483
 . PMID 19240804. doi:10.1371/journal.pone.0004603.
. PMID 19240804. doi:10.1371/journal.pone.0004603.
- ^ Knowles DG, McLysaght A. Recent de novo origin of human protein-coding genes. Genome Res. 2009, 19 (10): 1752–1759. PMC 2765279
 . PMID 19726446. doi:10.1101/gr.095026.109.
. PMID 19726446. doi:10.1101/gr.095026.109.
- ^ Wilson, Ben A.; Joanna Masel. Putatively Noncoding Transcripts Show Extensive Association with Ribosomes. Genome Biology & Evolution. 2011, 3: 1245–1252. doi:10.1093/gbe/evr099.
- ^ Tautz, Diethard and Domazet-Lošo, Tomislav. The evolutionary origin of orphan genes. Nature Reviews Genetics. 2011, 12 (10): 692–702. PMID 21878963. doi:10.1038/nrg3053.
- ^ Levine MT; Jones CD; Kern AD; et al. Novel genes derived from noncoding DNA in Drosophila melanogaster are frequently X-linked and exhibit testis-biased expression. Proc Natl Acad Sci USA. 2006, 103 (26): 9935–9939. Bibcode:2006PNAS..103.9935L. PMC 1502557
 . PMID 16777968. doi:10.1073/pnas.0509809103.
. PMID 16777968. doi:10.1073/pnas.0509809103.
- ^ Zhou Q; Zhang G; Zhang Y; et al. On the origin of new genes in Drosophila. Genome Res. 2008, 18 (9): 1446–1455. PMC 2527705
 . PMID 18550802. doi:10.1101/gr.076588.108.
. PMID 18550802. doi:10.1101/gr.076588.108.
- ^ Suárez-Díaz, Edna and Anaya-Muñoz, Victor H. History, objectivity, and the construction of molecular phylogenies. Stud. Hist. Phil. Biol. & Biomed. Sci. 2008, 39 (4): 451–468. PMID 19026976. doi:10.1016/j.shpsc.2008.09.002.
- ^ Erika Check Hayden. Technology: The $1,000 genome. Nature. 2014-03-20, 507: 294-295. doi:10.1038/507294a.
- ^ Lahr, D. J.; Laughinghouse, H. D.; Oliverio, A. M.; Gao, F.; Katz, L. A. How discordant morphological and molecular evolution among microorganisms can revise our notions of biodiversity on Earth. BioEssays. 2014, 36 (10): 950–959 [2015-07-16]. doi:10.1002/bies.201400056. (原始内容存档于2015-12-22).
- ^ Society for Molecular Biology and Evolution. [2015-07-16]. (原始内容存档于2016-03-08).
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Text is available under the CC BY-SA 4.0 license; additional terms may apply.
Images, videos and audio are available under their respective licenses.