動原体
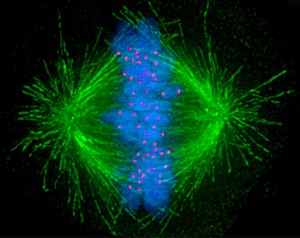
動原体(どうげんたい)またはキネトコア(英: kinetochore、[kɪˈnɛtəkɔːr]、[-ˈniːtəkɔːr])は、真核生物の細胞において、複製された染色分体に結合する円盤型のタンパク質構造であり、細胞分裂時に姉妹染色分体を引き離すために紡錘糸が結合する部位である[1]。キネトコアはセントロメア上で組み立てられ、有糸分裂と減数分裂時に染色体を紡錘体の微小管ポリマーへ連結する。また、キネトコアのタンパク質は姉妹染色分体をつなぎとめておくのを助け、染色体の編集にも関与する[2]。
脊椎動物、菌類、大部分の植物を含む、モノセントリック染色体の生物では、各染色体には1つのセントロメアが存在し、そこで1つのキネトコアが組み立てられる。線形動物や一部の植物などのホロセントリック染色体を持つ生物では、染色体全長にわたってキネトコアが組み立てられる[3]。
キネトコアは細胞分裂時の染色体の顕著な運動を開始し、制御し、監視する。S期に染色体が複製された後の有糸分裂の過程では、2つの姉妹染色分体はセントロメアによって保持されている。各染色分体にはそれぞれ反対方向をむいたキネトコアが存在しており、それぞれが紡錘体の各極へと連結される。中期から後期への移行後、姉妹染色分体は分離され、各染色体上のキネトコアが紡錘体極への移動を駆動する。2つの新たな娘細胞はこのようにして決定され、それゆえキネトコアは有糸分裂と減数分裂時の染色体分離に必要不可欠である。
構造
[編集]キネトコアは2つの領域から構成される。
- Inner kinetochoreと呼ばれる内側の領域はセントロメアDNAと堅く結合しており、細胞周期を通じて持続する特別なクロマチン構造へと組み立てられる。
- Outer kinetochoreと呼ばれる外側の領域は微小管と相互作用する。非常に動的な構造であり、細胞分裂時にのみ組み立てられて機能する。
最も単純なキネトコアであっても19種類以上の異なるタンパク質から構成される。こうしたタンパク質の多くは真核生物の間で保存されており、キネトコアのDNAへの結合を助ける特殊なヒストンH3のバリアント(CENP-AまたはCenH3と呼ばれる)などがある。キネトコアの他のタンパク質は紡錘体微小管への接着などを担っている。ダイニンやキネシンなどのモータータンパク質も含まれ、有糸分裂時の染色体の移動の動力を産生している。Mad2などは微小管との接着や姉妹キネトコア間の張力を監視し、これらのいずれかがみられない場合にはスピンドルチェックポイントを活性化して細胞周期を停止させる[4]。
キネトコアの機能には、紡錘体微小管への染色体の固定、固定の検証、スピンドルチェックポイントの活性化、染色体の移動の推進力の産生などがある[5]。微小管はα-チューブリンとβ-チューブリンからなる準安定性ポリマーであり、伸長と収縮を繰り返す。この現象は動的不安定性(dynamic instability)として知られている[6]。こうした非常に動的な性質はキネトコアの機能へと組み込まれ、染色体の移動と分離の制御が行われる。キネトコアの構成は有糸分裂時と減数分裂時で異なることも報告されており、相同染色体の対合、姉妹キネトコアの同一方向性(monoorientation)、セントロメアのコヒーシンの保護、スピンドル極体の結合と複製といった減数分裂特有のイベントには、減数分裂のために完全なキネトコア構成が必要不可欠である[7][8]。
動物細胞
[編集]キネトコアはいくつかの層で構成されており、こうした構造は電子顕微鏡を用いて観察されており、当初は従来型の固定染色法によって[9][10][11]、近年では迅速凍結置換法によって観察が行われている[12]。

キネトコアの最も深い層はinner plateである。ヌクレオソームからなるクロマチン構造上に構成されており、特殊なヒストン(この領域ではCENP-AがヒストンH3に置き換わる)、補助的タンパク質、DNAを含む。セントロメアのDNA構成(サテライトDNA)は、脊椎動物のキネトコアにおいて最も理解が進んでいない部分の1つである。Inner plateは、細胞周期を通じて分離されたヘテロクロマチンドメインのような外観をしている。
Inner plateの外部に位置しているのがouter plateであり、主にタンパク質で構成される。この構造は核膜が解体された後でのみ、染色体の表面に組み立てられる[9]。脊椎動物のキネトコアのouter plateには約20か所の微小管(+)端固定部位が存在するが、出芽酵母Saccharomyces cerevisiaeのouter plateには固定部位は1つしか存在しない。
キネトコアの最外部のドメインは繊維状のコロナを形成している。この構造は微小管が存在しないときにのみ、一般的な顕微鏡で可視化される。このコロナは常在型または一過的な動的タンパク質ネットワークによって形成されており、スピンドルチェックポイント、微小管固定、染色体移動の調節への関与が示唆されている。
有糸分裂の間、完全な染色体を形成している各姉妹染色分体にはそれぞれキネトコアが存在する。哺乳類の培養細胞では、明確な姉妹キネトコアはG2期の終わりに初めて観察される[13]。こうした初期キネトコアは、核膜が解体される前には成熟した層状構造を示すようになる[14]。

キネトコアで組み立てられる最初のタンパク質はCENP-A(出芽酵母ではCse4)である。このタンパク質はヒストンH3の特殊なアイソフォームである[15]。CENP-Aはinner kinetochoreのタンパク質CENP-C、CENP-H、CENP-I/MIS6の組み込みに必要である[16][17][18][19][20]。これらのタンパク質とCENP-A依存的経路との関係は完全には明確になっておらず、ニワトリ細胞ではCENP-Cの局在にはCENP-Hを必要し、ヒト細胞ではCENP-I/MIS6に非依存的である。線虫Caenorhabditis elegansと後生動物では、outer kinetochoreの多くのタンパク質は究極的にはCENP-Aに依存している。
キネトコアタンパク質は、有糸分裂時のキネトコアでの濃度によって分類することができる。一部のタンパク質は細胞分裂を通じて結合が維持されるが、他の一部のタンパク質の濃度は変化する。さらに、その結合部位でのリサイクルが迅速に行われる動的なタンパク質と、ゆるやかに交換が行われる安定なタンパク質がある。
- タンパク質のレベルが前期から後期まで安定したままのものとしては、inner plateの恒常的構成要素や、Ndc80複合体[21][22]、KNL/KBPタンパク質(kinetochore-null/KNL-binding protein)[23]、MISタンパク質[23]、CENP-F[24][25]などのouter plateの安定な構成要素がある。恒常的構成要素とともに、これらのタンパク質はキネトコアのコアを組織化しているようである。
- 有糸分裂時にキネトコア上の濃度が変動する動的な構成要素には、分子モーターCENP-Eとダイニン(とその標的の構成要素であるZW10とROD)、スピンドルチェックポイントタンパク質(Mad1、Mad2、BubR1、Cdc20など)がある。これらのタンパク質は微小管不在下で高濃度でキネトコア上に組み立てられるが、キネトコアに固定された微小管の数が多くなるほどこれらのタンパク質の濃度は低下する[26]。中期には、CENP-E、Bub3、Bub1のレベルは微小管が結合していないキネトコアと比較して1/3から1/4に低下するが、ダイニン/ダイナクチン、Mad1、Mad2、BubR1のレベルは1/10から1/100以下にまで低下する[26][27][28][29]。
- 微小管が固定されるとouter plateに存在するスピンドルチェックポイントタンパク質のレベルは低下するが[29]、EB1、APCなど他の構成要素や、Ran経路のタンパク質(RanGap1とRanBP2)は微小管が固定されているときにのみキネトコアと結合する[30][31][32][33]。これらは、キネトコアが微小管の(+)端を認識し、適切な固定を確保し、固定されたまま動的な挙動を調節する機構に関係していると考えられる。
2010年に、キネトコアを含む脊椎動物の染色体組成のプロテオミクス解析を目的として、multiclassifier combinatorial proteomics(MCCP)と名付けられた複雑な手法を用いた研究が行われた[34]。この研究では、キネトコアに対する生化学的濃縮過程は行われなかったにもかかわらず、得られたデータにはセントロメアの全てのサブ複合体を含む、既知の125種類のセントロメアタンパク質全てに由来するペプチドが含まれていた。この研究からは未知のキネトコアタンパク質が約100種類存在することが示され、有糸分裂時の既知構造の数は2倍となり、キネトコアが最も複雑な細胞内構造体の1つであることが確認された。また包括的な文献調査からは、既に少なくとも196種類のヒトタンパク質がキネトコアへの局在が実験的に示されていることが明らかにされた[35]。
機能
[編集]1つのキネトコアに対して接着する微小管の数はさまざまであり、出芽酵母では各キネトコアに1本の微小管が接着するだけであるが、哺乳類では各キネトコアに15–35本の微小管が接着する[36]。しかしながら、紡錘体の全ての微小管が1つのキネトコアに接着するのではない。1つの中心体から他の中心体へ伸びる微小管もあり、これらは紡錘体の長さを決定している。一部の短い微小管は長い微小管の間を結合している。微小管-キネトコア間の接着をレーザーによって切断すると、染色分体は移動することができず、異常な染色体分配が引き起こされる[37]。またこうした実験により、キネトコアには極性が存在し、キネトコアがどちらの中心体から伸びてきた微小管に接着するかはキネトコアの向きに依存していることが示された。この特異性により、各紡錘体極へ1つの染色分体だけが移動することが保証され、遺伝物質の正しい分配が保証されている。このように、キネトコアの基本的な機能の1つは紡錘体への接着であり、この過程は姉妹染色体の適切な分離に必要不可欠である。固定が不正確であった場合はエラーが生じ、染色体の異数性によって細胞には破滅的な結果が引き起こされる。これを防ぐため、エラーを検出して修正する機構が存在し、その構成要素はキネトコア上にも位置している。1つの染色分体が中心体へ向かう運動は主にキネトコアとの結合部位での微小管の脱重合によって生み出される。こうした運動には力の産生も必要であり、同様にキネトコア上に位置している分子モーターが関与している。
紡錘体微小管への染色体の固定
[編集]微小管の捕捉
[編集]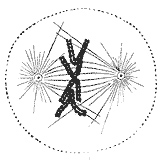
細胞周期のS期の間、中心体は複製を開始する。有糸分裂の開始段階には、各中心体の双方の中心小体の長さは最大となり、中心体はさらなる物質をリクルートして微小管の核形成能力が増大する。有糸分裂が進行するにつれて、紡錘体を形成するために双方の中心体は分離する[38]。このようにして、有糸分裂を行う細胞の紡錘体には微小管が伸びる2つの極が存在するようになる。微小管は非対称的な末端を持つ長いタンパク質性の繊維である。(−)端は比較的安定で中心体に隣接しており、(+)端は伸長と短縮を交互に繰り返しながら細胞の中心部を探索する。この探索過程で、微小管は染色体と遭遇し、キネトコアを介して捕捉する[39][40]。キネトコアを発見し接着した微小管は安定化されるが、遊離したままの微小管は迅速に脱重合される[41]。染色体には2つのキネトコアが背中合わせに(各姉妹染色分体に1つずつ)結合しているため、一方のキネトコアが1つの極から形成された微小管と接着すると、姉妹染色分体に存在する他方のキネトコアは反対側の極を向くこととなる。そのためほとんどの場合、2つ目のキネトコアは反対側の極から伸びてきた微小管と結合する[42]。このようにして染色体は二方向性(bi-oriented)またはアンフィテリック(amphitelic)と呼ばれる状態となり、この基本的配置によって細胞が分裂した際の双方の染色分体の適切な分離が保証される[43][44]。
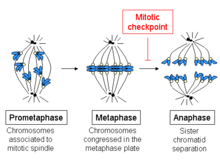
1つのキネトコアに1つの微小管が固定されると、結合した染色体が極に向かう運動が迅速に開始される。この運動はおそらく、微小管に固定されていないキネトコアに濃縮されて存在しているモータータンパク質ダイニンが(−)端へ向かう運動によって媒介されている[45][46][47]。ダイニンはキネトコア微小管(キネトコアに固定された微小管)が結合すると放出される[26]。極へ向かう運動はキネトコアにキネトコア微小管が結合している限り緩やかなものとなり、運動はキネトコア微小管の長さの変化によって行われるようになる。培養哺乳類細胞では、ダイニンはスピンドルチェックポイントの不活性化には必要であるが、紡錘体の赤道面への集合やキネトコア微小管の獲得、後期の染色体分離には不要である[48]。高等植物や酵母にダイニンが存在する証拠はないが、(−)端へ向かう他のキネシンがダイニンの不在を補っていると考えられている。

微小管の初期の捕捉に関与している他のモータータンパク質はCENP-Eである。CENP-Eは高分子量キネシンで、哺乳類のキネトコアで前中期から後期まで繊維状のコロナと結合している[49]。CENP-Eのレベルが低い細胞では染色体のキネトコアにこのタンパク質が存在せず、かなりの頻度で中期板へ集合する能力に欠陥が生じる。こうしたケースでは、大部分の染色体は正しく中期板に集合するが、一部の染色体は慢性的にmono-oriented状態(一方の極のみから固定された状態)のままとなる[50]。
キネトコア微小管は中心小体、哺乳類培養細胞では紡錘体極で重合した微小管に由来するものであることは広く受け入れられている[39]。一方で、キネトコアで直接重合した微小管が大きく寄与している可能性もある[51]。
Ndc80複合体の役割
[編集]キネトコアに結合した微小管は特別な特徴を示す。遊離している微小管と比較して、キネトコア微小管は低温による脱重合、高い静水圧、カルシウムへの曝露に対してかなり高い抵抗性を示す[52]。さらに、キネトコア微小管は遊離(+)端を持つ星状体微小管や紡錘体微小管よりも再生がかなり遅いが、レーザーによってキネトコアから解離した場合には迅速に脱重合する[37]。
ダイニンやCENP-Eはキネトコア微小管の形成に必要不可欠ではなく、キネトコア微小管の固定を担っているのはNdc80複合体であることが酵母での先駆的研究により明らかにされている[21][53][54][55]。出芽酵母では、Ndc80複合体は、Ndc80p、Nuf2p、Spc24p、Spc25pの4つの構成要素からなる。この複合体のいずれかの構成要素を欠失した変異体では、キネトコアの構造が完全に失われるわけではないが、キネトコアと微小管の連結が失われる[21][53]。キネトコアの構造が失われる変異体(酵母のNdc10の変異体など[56])では、微小管との連結とスピンドルチェックポイントの活性化の双方が失われる。これは、キネトコアがスピンドルチェックポイント構成要素が組み立てられるプラットフォームとして機能しているためであると考えられる。
Ndc80複合体は高度に保存されており、分裂酵母Schizosaccharomyces pombe、線虫Caenorhabditis elegans、ツメガエルXenopus、ニワトリ、ヒトでも同定されている[21][22][53][57][58][59][60]。Ndc80pのヒトホモログであるHec1の研究からは、適切な染色体の集合と有糸分裂の進行に重要であること、そしてコヒーシンやコンデンシン複合体の構成要素と相互作用することが示されている[61]。
さまざまな研究によって、Ndc80複合体はキネトコア-微小管間の固定の安定化に必要不可欠であり、高等真核生物での適切な染色体集合の確立へ関与していることが示唆されている、セントロメアでの張力の発生の補助に必要であることが示されている[22][58][59][60]。RNAiや遺伝子ノックアウト、抗体のマイクロインジェクションを用いてNdc80の機能不全となった細胞では、異常に長い紡錘体が形成され、姉妹キネトコア間の張力が失われ、染色体は中期板に集合することができなくなり、キネトコア微小管の結合はほとんど見られなくなる。
Ndc80複合体が直接微小管と結合し、キネトコア-微小管間相互作用面の核となる構成要素を形成していることを支持する、さまざまな強力な証拠が存在する[62]。しかしながら、堅固なキネトコア-微小管間相互作用の形成にはさらなるタンパク質の機能が必要である可能性がある。酵母では、この連結にはDam1-DASH-DDD複合体の存在が必要である。この複合体の一部のメンバーは微小管に直接結合するが、他の因子はNdc80複合体に結合する[54][55][63]。このことは、Dam1-DASH-DDD複合体はキネトコアと微小管の間の必要不可欠なアダプターである可能性を意味している。しかしながら、動物では対応する複合体は同定されておらず、この疑問に対して現在精力的な研究が行われている。
動原体-微小管結合の検証
[編集]S期の間に、細胞は染色体に保存されているすべての遺伝情報を複製し、この過程はDNA複製と呼ばれる。この過程の終わりには、各染色体には姉妹染色分体が含まれ、この2つの染色分体は完全で同一なDNA分子である。2つの染色分体は、染色体分離が起こる後期までコヒーシン複合体によって結合したままである。染色体分離が正しく起こると、各娘細胞は染色分体の完全なセットを受け取る。そのためには、各姉妹染色分体はそれぞれ紡錘体の反対側の極で形成された微小管へと固定されている必要がある。この構成はアンフィテリック(amphitelic)または二方向性(bi-orientation)と呼ばれている。しかし、固定の過程で正しくない構成が生じることもある[64]。

- モノテリック(monotelic): 染色分体の1つだけが微小管に固定されており、2つ目のキネトコアは固定されていない。この場合、セントロメアで張力が発生しないためスピンドルチェックポイントが活性化され、後期の開始が遅れてエラーの修正が行われる。修正が行われなかった場合には、固定されていない染色分体は2つの娘細胞のどちらかにランダムに移動して染色体異数性が生じる。すなわち、一方の娘細胞には過剰な染色体が、他方では染色体の不足が生じることとなる。
- シンテリック(syntelic): 双方の染色分体が同じ極からの微小管に固定される。この場合もセントロメアの張力が生み出されず、スピンドルチェックポイントが活性化される。修正されなかった場合、双方の染色分体が同じ娘細胞へ移動し、異数性が生じる。
- メロテリック(merotelic): 少なくとも1つの染色分体が同時に双方の極からの微小管に固定されている。この場合はセントロメアの張力が生み出されるため、スピンドルチェックポイントは活性化されない。修正されなかった場合、双方の極に結合している染色分体は後期に中央部に取り残され、最終的には2つの断片へと破壊さて娘細胞へ分配され、異数性が生じる。
モノテリック型とシンテリック型の配置ではセントロメアの張力が生み出されないため、スピンドルチェックポイントによって検出される。対照的に、メロテリック型配置はこの制御機構では検出されない。しかし、こうしたエラーの大部分は細胞が後期に入るまでに検出されて修正される[64]。こうした固定のエラーの修正に重要な因子は染色体パッセンジャー複合体(chromosomal passenger complex)であり、オーロラBキナーゼ、その標的かつ活性化サブユニットであるINCENP、他の2つのサブユニットサバイビンとBorealin/Dasra B(CDCA8)が含まれる[65]。ドミナントネガティブ変異体、RNAi、抗体マイクロインジェクションや選択的薬剤を用いてこの複合体の機能を破壊した細胞では、染色体固定のエラーが蓄積する。多くの研究によって、オーロラBは不正確なキネトコア-微小管固定の不安定化に必要であり、アンフィテリック型の結合の形成を促進することが示されている。オーロラBの酵母ホモログ(Ipl1p)は、キネトコアの構成的タンパク質であるNdc10pやNdc80複合体、Dam1-DASH-DDD複合体のメンバーなどをリン酸化する[66]。Ndc80複合体の構成要素のリン酸化は、キネトコア微小管固定の不安定化を引き起こす。その機能にはオーロラBの局在が重要であることが提唱されている。オーロラBはキネトコアの内部領域(セントロメアヘテロクロマチン)に局在しているため、セントロメアの張力が確立されて姉妹キネトコアが分離された際にはオーロラBはその基質に到達することができず、その結果キネトコア微小管は安定化される。オーロラBはいくつかのがんのタイプで高頻度で過剰発現しており、現在抗がん剤の開発の標的となっている[67]。
スピンドルチェックポイントの活性化
[編集]スピンドル(紡錘体)チェックポイントまたはスピンドル形成チェックポイント(spindle assembly checkpoint、SAC)は、有糸分裂チェックポイント(mitotic checkpoint)とも呼ばれ、
- 有糸分裂紡錘体が正しく組み立てられていること
- 全ての染色体が紡錘体の双方の極と接着していること
- 全ての染色体が中期板に集合していること
の検知を担う。集合過程で染色体が(いかなる理由であれ)1本でも取り残されている場合には、スピンドルチェックポイントは細胞周期の進行を遅らせる。この停止期間の間に、細胞は修復機構によって検知された問題を解消する。ある程度の時間の後も問題が解消されない場合は、細胞はアポトーシス(プログラム細胞死)の標的となる。アポトーシスは、染色体異数性を避けるための安全機構である。
CENP-Bなどの構造的セントロメアタンパク質は有糸分裂を通じて(終期まで)安定して局在したままであるのに対し、スピンドルチェックポイントの構成要素は微小管不在下に高濃度でキネトコアで組み立てられ、キネトコアに接着した微小管の数が増加するにつれてその濃度は低下する[26]。
中期には、CENP-E、Bub3、Bub1のレベルは未接着のキネトコアと比較して1/3から1/4に低下し、ダイニン/ダイナクチン、Mad1、Mad2、BubR1のレベルは1/10から1/100に低下する[26][27][28][29]。中期にすべての染色体が中期板に整列した場合には、すべてのチェックポイントタンパク質がキネトコアから放出される。チェックポイントタンパク質がキネトコアから消失したことは、染色体が中期板に到達し、双方向からの張力が発生していることを示している。この瞬間にCdc20に結合して阻害しているチェックポイントタンパク質(Mad1-Mad2とBubR1)がCdc20を放出し、Cdc20はAPC/Cに結合してAPC/CCdc20となって活性化する。この複合体は姉妹染色分体の分離を開始させ、後期への進行を引き起こす。
いくつかの研究では、Ndc80複合体がMad1-Mad2とダイニンのキネトコアへの安定な結合の調節に関与していることが示唆されている[22][59][60]。一方で、キネトコア結合タンパク質CENP-A、CENP-C、CENP-E、CENP-H、BubR1はNdc80/Hec1には依存していない。Ndc80/Hec1のレベルが低い細胞で観察される前中期での停止の延長はMad2に依存しているが、これらの細胞ではキネトコア上のMad1、Mad2、ダイニンのレベルが低下する。Ndc80/Hec1とNuf2のレベルが共に低下している場合には、Mad1とMad2はキネトコアから完全に消失し、スピンドルチェックポイントは不活性化される[68]。
シュゴシン(Sgo1、キイロショウジョウバエDrosophila melanogasterではMEI-S332[69])は、コヒーシンが後期までセントロメアに結合した状態を維持するために必要不可欠なセントロメアタンパク質である。ヒトのSgo1は前期にセントロメアに結合し、後期の開始時に消失する[70]。HeLa細胞においてシュゴシンのレベルをRNAiによって低下させると、コヒーシンは有糸分裂時にセントロメアにとどまることができなくなり、姉妹染色分体は後期が開始される前に分離し、有糸分裂の長期間の停止が引き起こされる。
また、Ranサイクルに関与するタンパク質(RanGAP1とRanBP2/Nup358)が有糸分裂時のキネトコアでは検出される。RanGAP1はGTP結合型RanからGDP結合型Ranへの変換を促進するGTPアーゼ活性化タンパク質で、RanBP2はRan結合タンパク質である[71]。これらのタンパク質は間期には核膜孔に位置し、核-細胞質間輸送に関与している。GTP結合型Ranのレベルを増加させる処理によってBub1、Bub3、Mad2、CENP-Eのキネトコアからの解離が阻害されることから、これらのタンパク質のキネトコアへの局在には機能的意義があるようである[72]。
さらに、S期のDNA複製の開始に関与している複製起点認識複合体(ORC)のタンパク質Orc2もヒト細胞では有糸分裂時にキネトコアに局在している[73]。このことは、一部の研究で酵母のOrc2が姉妹染色分体の結合に関与しており、Orc2が細胞からの除去によってスピンドルチェックポイントが活性化することが示唆されていることと一致する[74]。他のORCの構成要素(分裂酵母のorc5など)も結合に関与していることが判明している[75]。
染色体の移動を推進する力の産生
[編集]紡錘体極に関係した染色体の運動の大部分は、キネトコア微小管の伸長や短縮と関係したものである。キネトコアは、結合した(約20本の)キネトコア微小管の(+)端を脱重合状態から重合状態へ変更することができる。これによって前中期の細胞のキネトコアは"directional instability"[76]と呼ばれる状態となり、極方向への運動と逆方向への運動という持続的な段階の間を移行する。これらの運動はキネトコア微小管の脱重合状態、重合状態とそれぞれ共役している。こうしたキネトコアの双安定性は、キネトコアと紡錘体極との機械的連結を失うことなく、染色体を紡錘体の赤道面に整列させる機構の一部をなしているようである。キネトコアの双安定性はキネトコア微小管の(+)端の動的不安定性に基づいており、部分的にはキネトコアにおける張力の存在によって制御されている。哺乳類の培養細胞では、キネトコアでの張力が低い場合にはキネトコア微小管が脱重合する方向の変化が促進され、高い場合にはキネトコア微小管が重合する方向の変化が促進される[77][78]。
キネトコアタンパク質と微小管の(+)端に結合するタンパク質(まとめて+TIPsと呼ばれる)は、キネトコア微小管の(+)端のダイナミクスの調節によってキネトコアの運動を調節する[79]。キネトコア-微小管間の相互作用面は高度に動的であり、これらのタンパク質の一部は双方の構造の構成要素となっているようである。KinIなどのキネシンはデポリメラーゼ(脱重合酵素)のように機能する。微小管の(+)端に結合する+TIPsは重合を促進し、デポリメラーゼ作用に対抗すると考えられている[80]。
- KinIキネシンは、モータードメインが配列の内部(internal)にそんざいするためKin"I"と名付けられている。ATPを利用してチューブリンポリマー(微小管)の脱重合を促進する。脊椎動物では、(+)端の重合のダイナミクスを制御する最も重要なKinIキネシンはMCAKである[81]。一方で、他のキネシンも関与しているようである。
- キネトコアでの機能をの持つ+TIPsには2つのグループがある。
- 1つ目グループはAPCタンパク質とその結合タンパク質EB1を含み、微小管がキネトコアに局在するために必要である。どちらのタンパク質も適切な染色体分離に必要である[82]。EB1は重合状態の微小管にのみ結合し、キネトコア微小管の安定化を促進していることが示唆される。
- 2つ目のグループは微小管が存在しなくてもキネトコアに局在することができる。このグループでは、CLIP-170とCLASPs(CLIP-associated proteins)が広く研究されている。CLIP-170のキネトコアでの役割は不明であるが、CLIP-170のドミナントネガティブ型変異体の発現は前中期の遅延を引き起こすことから[83]、染色体の整列に活発な役割を有していることが示唆される。ヒト、ショウジョウバエ、酵母において、CLASPsは染色体の整列と双極型の紡錘体の維持に必要である[84][85]。
出典
[編集]- ^ Santaguida, Stefano; Musacchio, Andrea (2009-09-02). “The life and miracles of kinetochores”. The EMBO Journal 28 (17): 2511–2531. doi:10.1038/emboj.2009.173. ISSN 1460-2075. PMC 2722247. PMID 19629042.
- ^ Brooker, Robert J. (2016). Concepts of Genetics. New York: McGraw Hill Education
- ^ Albertson, D.G.; Thomson, J.N. (1993), “Segregation of holocentric chromosomes at meiosis in the nematode, Caenorhabditis elegans”, Chromosome Research 1 (1): 15–26, doi:10.1007/BF00710603, PMID 8143084
- ^ Peter De Wulf, William C. Earnshaw, The Kinetochore: From Molecular Discoveries to Cancer Therapy
- ^ a b Maiato, H.; Deluca, J.; Salmon, E.D.; Earnshaw, W.C. (2004), “The dynamic kinetochore-microtubule interface”, Journal of Cell Science 117 (22): 5461–5477, doi:10.1242/jcs.01536, PMID 15509863
- ^ Mitchison, T.; Kirschner, M. (1984), “Dynamic instability of microtubule growth”, Nature 312 (5991): 237–242, doi:10.1038/312237a0, PMID 6504138, オリジナルの2010-06-22時点におけるアーカイブ。 2010年8月23日閲覧。
- ^ Mehta, G.D.; Agarwal, M.; Ghosh, S.K. (2014), “Functional characterization of kinetochore protein, Ctf19 in meiosis I: an implication of differential impact of Ctf19 on the assembly of mitotic and meiotic kinetochores in Saccharomyces cerevisiae”, Molecular Microbiology 91 (6): 1179–1199, doi:10.1111/mmi.12527, PMID 24446862
- ^ Agarwal, Meenakshi; Mehta, Gunjan; Ghosh, Santanu K. (2015-03-01). “Role of Ctf3 and COMA subcomplexes in meiosis: Implication in maintaining Cse4 at the centromere and numeric spindle poles”. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research 1853 (3): 671–684. doi:10.1016/j.bbamcr.2014.12.032. ISSN 0167-4889. PMID 25562757.
- ^ a b Brinkley, B.R.; Stubblefield, E. (1966), “The fine structure of the kinetochore of a mammalian cell in vitro”, Chromosoma 19 (1): 28–43, doi:10.1007/BF00332792, PMID 5912064
- ^ Jokelainen, P.T. (1967), “The ultrastructure and spatial organization of the metaphase kinetochore in mitotic rat cells”, J Ultrastruct Res 19 (1): 19–44, doi:10.1016/S0022-5320(67)80058-3, PMID 5339062
- ^ Rieder, C.L. (1982), “The formation, structure, and composition of the mammalian kinetochore and kinetochore fiber”, Int Rev Cytol, International Review of Cytology 79: 1–58, doi:10.1016/S0074-7696(08)61672-1, ISBN 978-0-12-364479-4, PMID 6185450
- ^ McEwen, B.F.; Hsieh, C.E.; Mattheyses, A.L.; Rieder, C.L. (1998), “A new look at kinetochore structure in vertebrate somatic cells using high-pressure freezing and freeze substitution”, Chromosoma 107 (6): 366–375, doi:10.1007/s004120050320, PMC 2905855, PMID 9914368
- ^ Brenner, S.; Pepper, D.; Berns, M.W.; Tan, E.; Brinkley, B.R. (1981), “Kinetochore structure, duplication, and distribution in mammalian cells: analysis by human autoantibodies from scleroderma patients”, The Journal of Cell Biology 91 (1): 95–102, doi:10.1083/jcb.91.1.95, PMC 2111947, PMID 7298727
- ^ Pluta, A.F.; MacKay, A.M.; Ainsztein, A.M.; Goldberg, I.G.; Earnshaw, W.C. (1995), “The Centromere: Hub of Chromosomal Activities”, Science 270 (5242): 1591–4, doi:10.1126/science.270.5242.1591, PMID 7502067
- ^ Palmer, D.K.; O'Day, K.; Trong, H.L.; Charbonneau, H.; Margolis, R.L. (1991), “Purification of the centromere-specific protein CENP-A and demonstration that it is a distinctive histone”, Proceedings of the National Academy of Sciences 88 (9): 3734–3738, doi:10.1073/pnas.88.9.3734, PMC 51527, PMID 2023923
- ^ Howman, E.V.; Fowler, K.J.; Newson, A.J.; Redward, S.; MacDonald, A.C.; Kalitsis, P.; Choo, K.H.A. (2000), “Early disruption of centromeric chromatin organization in centromere protein A (Cenpa) null mice”, Proceedings of the National Academy of Sciences 97 (3): 1148–1153, doi:10.1073/pnas.97.3.1148, PMC 15551, PMID 10655499
- ^ Oegema, K.; Desai, A.; Rybina, S.; Kirkham, M.; Hyman, A.A. (2001), “Functional Analysis of Kinetochore Assembly in Caenorhabditis elegans”, The Journal of Cell Biology 153 (6): 1209–1226, doi:10.1083/jcb.153.6.1209, PMC 2192036, PMID 11402065
- ^ Van Hooser, A.A.; Ouspenski, I.I.; Gregson, H.C.; Starr, D.A.; Yen, T.J.; Goldberg, M.L.; Yokomori, K.; Earnshaw, W.C. et al. (2001), “Specification of kinetochore-forming chromatin by the histone H3 variant CENP-A”, Journal of Cell Science 114 (19): 3529–3542, PMID 11682612
- ^ Fukagawa, T.; Mikami, Y.; Nishihashi, A.; Regnier, V.; Haraguchi, T.; Hiraoka, Y.; Sugata, N.; Todokoro, K. et al. (2001), “CENP-H, a constitutive centromere component, is required for centromere targeting of CENP-C in vertebrate cells”, The EMBO Journal 20 (16): 4603–4617, doi:10.1093/emboj/20.16.4603, PMC 125570, PMID 11500386
- ^ Goshima, G.; Kiyomitsu, T.; Yoda, K.; Yanagida, M. (2003), “Human centromere chromatin protein hMis12, essential for equal segregation, is independent of CENP-A loading pathway”, The Journal of Cell Biology 160 (1): 25–39, doi:10.1083/jcb.200210005, PMC 2172742, PMID 12515822
- ^ a b c d Wigge, Philip A.; Kilmartin, John V. (2001), “The Ndc80p Complex from Saccharomyces cerevisiae Contains Conserved Centromere Components and Has a Function in Chromosome Segregation”, The Journal of Cell Biology 152 (2): 349–360, doi:10.1083/jcb.152.2.349, PMC 2199619, PMID 11266451
- ^ a b c d Deluca, J.G.; Moree, B.; Hickey, J.M.; Kilmartin, J.V.; Salmon, E.D. (2002), “hNuf2 inhibition blocks stable kinetochore–microtubule attachment and induces mitotic cell death in HeLa cells”, The Journal of Cell Biology 159 (4): 549–555, doi:10.1083/jcb.200208159, PMC 2173110, PMID 12438418
- ^ a b Cheeseman, I.M.; Niessen, S.; Anderson, S.; Hyndman, F.; Yates, J.R.; Oegema, K.; Desai, A. (2004), “A conserved protein network controls assembly of the outer kinetochore and its ability to sustain tension”, Genes & Development 18 (18): 2255–2268, doi:10.1101/gad.1234104, PMC 517519, PMID 15371340
- ^ Rattner, J.B.; Rao, A.; Fritzler, M.J.; Valencia, D.W.; Yen, T.J. (1993), “CENP-F is a. Ca 400 kDa kinetochore protein that exhibits a cell-cycle dependent localization”, Cell Motil Cytoskeleton 26 (3): 214–26, doi:10.1002/cm.970260305, PMID 7904902
- ^ Liao, H.; Winkfein, RJ; Mack, G; Rattner, JB; Yen, TJ (1995), “CENP-F is a protein of the nuclear matrix that assembles onto kinetochores at late G2 and is rapidly degraded after mitosis”, The Journal of Cell Biology 130 (3): 507–518, doi:10.1083/jcb.130.3.507, PMC 2120529, PMID 7542657
- ^ a b c d e Hoffman, DB; Hoffman, D.B.; Pearson, C.G.; Yen, T.J.; Howell, B.J.; Salmon, E.D. (2001), “Microtubule-dependent changes in assembly of microtubule motor proteins and mitotic spindle checkpoint proteins at PtK1 kinetochores”, Molecular Biology of the Cell 12 (7): 1995–2009, doi:10.1091/mbc.12.7.1995, PMC 55648, PMID 11451998
- ^ a b King, S.M. (2000), “The dynein microtubule motor”, Biochimica et Biophysica Acta (BBA) - Molecular Cell Research 1496 (1): 60–75, doi:10.1016/S0167-4889(00)00009-4, PMID 10722877
- ^ a b Howell, B.J.; Moree, B.; Farrar, E.M.; Stewart, S.; Fang, G.; Salmon, E.D. (2004), “Spindle Checkpoint Protein Dynamics at Kinetochores in Living Cells”, Current Biology 14 (11): 953–964, doi:10.1016/j.cub.2004.05.053, PMID 15182668
- ^ a b c Shah, J.V.; Botvinick, E.; Bonday, Z.; Furnari, F.; Berns, M.; Cleveland, D.W. (2004), “Dynamics of Centromere and Kinetochore Proteins Implications for Checkpoint Signaling and Silencing”, Current Biology 14 (11): 942–952, doi:10.1016/j.cub.2004.05.046, PMID 15182667
- ^ Tirnauer, Jennifer S.; Canman, Julie C.; Salmon, E.D.; Mitchison, Timothy J. (2002), “EB1 Targets to Kinetochores with Attached, Polymerizing Microtubules”, Molecular Biology of the Cell 13 (12): 4308–4316, doi:10.1091/mbc.E02-04-0236, PMC 138635, PMID 12475954
- ^ Kaplan, K.B.; Burds, A.A.; Swedlow, J.R.; Bekir, S.S.; Sorger, P.K.; Näthke, I.S. (2001), “A role for the Adenomatous Polyposis Coli protein in chromosome segregation”, Nature Cell Biology 3 (4): 429–432, doi:10.1038/35070123, PMID 11283619
- ^ Joseph, J.; Liu, S.T.; Jablonski, S.A.; Yen, T.J.; Dasso, M. (2004), “The RanGAP1-RanBP2 Complex is Essential for Microtubule-Kinetochore Interactions in Vivo”, Current Biology 14 (7): 611–617, doi:10.1016/j.cub.2004.03.031, PMID 15062103
- ^ Salina, Davide; Enarson, Paul; Rattner, J.B.; Burke, Brian (2003), “Nup358 integrates nuclear envelope breakdown with kinetochore assembly”, The Journal of Cell Biology 162 (6): 991–1002, doi:10.1083/jcb.200304080, PMC 2172838, PMID 12963708
- ^ “The Protein Composition of Mitotic Chromosomes Determined Using Multiclassifier Combinatorial Proteomics”, Cell 142 (5): 810–21, (September 2010), doi:10.1016/j.cell.2010.07.047, PMC 2982257, PMID 20813266
- ^ “Identification of novel mitosis regulators through data mining with human centromere/kinetochore proteins as group queries”, BMC Cell Biol 13: 15, (2012), doi:10.1186/1471-2121-13-15, PMC 3419070, PMID 22712476
- ^ McEwen, B.F.; Heagle, A.B.; Cassels, G.O.; Buttle, K.F.; Rieder, C.L. (1997), “Kinetochore Fiber Maturation in PtK1 Cells and Its Implications for the Mechanisms of Chromosome Congression and Anaphase Onset”, The Journal of Cell Biology 137 (7): 1567–1580, doi:10.1083/jcb.137.7.1567, PMC 2137823, PMID 9199171
- ^ a b Nicklas, R.B.; Kubai, D.F. (1985), “Microtubules, chromosome movement, and reorientation after chromosomes are detached from the spindle by micromanipulation”, Chromosoma 92 (4): 313–324, doi:10.1007/BF00329815, PMID 4042772
- ^ Mayor, T.; Meraldi, P.; Stierhof, Y.D.; Nigg, E.A.; Fry, A.M. (1999), “Protein kinases in control of the centrosome cycle”, FEBS Letters 452 (1–2): 92–95, doi:10.1016/S0014-5793(99)00534-7, PMID 10376685
- ^ a b Kirschner, M.; Mitchison, T. (1986), “Beyond self-assembly: from microtubules to morphogenesis”, Cell 45 (3): 329–342, doi:10.1016/0092-8674(86)90318-1, PMID 3516413
- ^ Holy, T. E.; Leibler, S. (1994), “Dynamic instability of microtubules as an efficient way to search in space”, Proceedings of the National Academy of Sciences of the United States of America 91 (12): 5682–5685, doi:10.1073/pnas.91.12.5682, PMC 44060, PMID 8202548
- ^ Hayden, J.H.; Bowser, SS; Rieder, CL (1990), “Kinetochores capture astral microtubules during chromosome attachment to the mitotic spindle: direct visualization in live newt lung cells”, The Journal of Cell Biology 111 (3): 1039–1045, doi:10.1083/jcb.111.3.1039, PMC 2116290, PMID 2391359
- ^ Nicklas, R.B. (1997), “How Cells Get the Right Chromosomes”, Science 275 (5300): 632–7, doi:10.1126/science.275.5300.632, PMID 9005842
- ^ Loncarek, J.; Kisurina-evgenieva, O.; Vinogradova, T.; Hergert, P.; La Terra, S.; Kapoor, T.M.; Khodjakov, A. (2007), “The centromere geometry essential for error-free mitosis is controlled by spindle forces”, Nature 450 (7170): 745–9, doi:10.1038/nature06344, PMC 2586812, PMID 18046416
- ^ Dewar, H.; Tanaka, K.; Nasmyth, K.; Tanaka, T.U. (2004), “Tension between two kinetochores suffices for their bi-orientation on the mitotic spindle”, Nature 428 (6978): 93–7, doi:10.1038/nature02328, PMID 14961024
- ^ Echeverri, C.J.; Paschal, B.M.; Vaughan, K.T.; Vallee, R.B. (1996), “Molecular characterization of the 50-kD subunit of dynactin reveals function for the complex in chromosome alignment and spindle organization during mitosis”, The Journal of Cell Biology 132 (4): 617–633, doi:10.1083/jcb.132.4.617, PMC 2199864, PMID 8647893
- ^ Sharp, D.J.; Rogers, G.C.; Scholey, J.M. (2000), “Cytoplasmic dynein is required for poleward chromosome movement during mitosis in Drosophila embryos”, Nature Cell Biology 2 (12): 922–930, doi:10.1038/35046574, PMID 11146657
- ^ Banks, J.D.; Heald, R. (2001), “Chromosome movement: Dynein-out at the kinetochore”, Current Biology 11 (4): 128–131, doi:10.1016/S0960-9822(01)00059-8, PMID 11250166
- ^ Howell, B.J.; McEwen, B.F.; Canman, J.C.; Hoffman, D.B.; Farrar, E.M.; Rieder, C.L.; Salmon, E.D. (2001), “Cytoplasmic dynein/dynactin drives kinetochore protein transport to the spindle poles and has a role in mitotic spindle checkpoint inactivation”, The Journal of Cell Biology 155 (7): 1159–1172, doi:10.1083/jcb.200105093, PMC 2199338, PMID 11756470
- ^ Cooke, C.A.; Schaar, B.; Yen, T.J.; Earnshaw, W.C. (1997), “LLocalization of CENP-E in the fibrous corona and outer plate of mammalian kinetochores from prometaphase through anaphase”, Chromosoma 106 (7): 446–455, doi:10.1007/s004120050266, PMID 9391217
- ^ Weaver, Beth A.A.; Bonday, Zahid Q.; Putkey, Frances R.; Kops, Geert J.P.L.; Silk, Alain D.; Cleveland, Don W. (2003), “Centromere-associated protein-E is essential for the mammalian mitotic checkpoint to prevent aneuploidy due to single chromosome loss”, The Journal of Cell Biology 162 (4): 551–563, doi:10.1083/jcb.200303167, PMC 2173788, PMID 12925705
- ^ a b Maiato, H.; Rieder, C.L.; Khodjakov, A. (2004), “Kinetochore-driven formation of kinetochore fibers contributes to spindle assembly during animal mitosis”, The Journal of Cell Biology 167 (5): 831–840, doi:10.1083/jcb.200407090, PMC 2172442, PMID 15569709
- ^ Mitchison, T.J. (1988), “Microtubule Dynamics and Kinetochore Function in Mitosis”, Annual Review of Cell Biology 4 (1): 527–545, doi:10.1146/annurev.cb.04.110188.002523, PMID 3058165
- ^ a b c He, X.; Rines, D.R.; Espelin, C.W.; Sorger, P.K. (2001), “Molecular Analysis of Kinetochore-Microtubule Attachment in Budding Yeast”, Cell 106 (2): 195–206, doi:10.1016/S0092-8674(01)00438-X, PMID 11511347
- ^ a b Westermann, Stefan; Cheeseman, Iain M.; Anderson, Scott; Yates, John R.; I. I. I., DG; Drubin, David G.; Barnes, Georjana (2003), “Architecture of the budding yeast kinetochore reveals a conserved molecular core”, The Journal of Cell Biology 163 (2): 215–22, doi:10.1083/jcb.200305100, PMC 2173538, PMID 14581449
- ^ a b De Wulf, P.; McAinsh, A.D.; Sorger, P.K. (2003), “Hierarchical assembly of the budding yeast kinetochore from multiple subcomplexes”, Genes & Development 17 (23): 2902–2921, doi:10.1101/gad.1144403, PMC 289150, PMID 14633972
- ^ Goh, P.Y.; Kilmartin, J.V. (1993), “NDC10: a gene involved in chromosome segregation in Saccharomyces cerevisiae”, The Journal of Cell Biology 121 (3): 503–12, doi:10.1083/jcb.121.3.503, PMC 2119568, PMID 8486732
- ^ Nabetani, A.; Koujin, T.; Tsutsumi, C.; Haraguchi, T.; Hiraoka, Y. (2001), “A conserved protein, Nuf2, is implicated in connecting the centromere to the spindle during chromosome segregation: a link between the kinetochore function and the spindle checkpoint”, Chromosoma 110 (5): 322–334, doi:10.1007/s004120100153, PMID 11685532
- ^ a b Howe, Mary; McDonald, Kent L.; Albertson, Donna G.; Meyer, Barbara J. (2001), “Him-10 Is Required for Kinetochore Structure and Function on Caenorhabditis elegans Holocentric Chromosomes”, The Journal of Cell Biology 153 (6): 1227–1238, doi:10.1083/jcb.153.6.1227, PMC 2192032, PMID 11402066
- ^ a b c Martin-lluesma, Silvia; Stucke, Volker M.; Nigg, Erich A. (2002), “Role of Hec1 in Spindle Checkpoint Signaling and Kinetochore Recruitment of Mad1/Mad2”, Science 297 (5590): 2267–2270, doi:10.1126/science.1075596, PMID 12351790
- ^ a b c McCleland, M.L.; Gardner, R.D.; Kallio, M.J.; Daum, J.R.; Gorbsky, G.J.; Burke, D.J.; Stukenberg, P.T. (2003), “The highly conserved Ndc80 complex is required for kinetochore assembly, chromosome congression, and spindle checkpoint activity”, Genes & Development 17 (1): 101–114, doi:10.1101/gad.1040903, PMC 195965, PMID 12514103
- ^ Zheng, L.; Chen, Y.; Lee, W.H. (1999), “Hec1p, an Evolutionarily Conserved Coiled-Coil Protein, Modulates Chromosome Segregation through Interaction with SMC Proteins”, Molecular and Cellular Biology 19 (8): 5417–5428, doi:10.1128/mcb.19.8.5417, PMC 84384, PMID 10409732
- ^ Wei, Ronnie R.; Al-bassam, Jawdat; Harrison, Stephen C. (2007), “The Ndc80/HEC1 complex is a contact point for kinetochore-microtubule attachment”, Nature Structural & Molecular Biology 14 (1): 54–59, doi:10.1038/nsmb1186, PMID 17195848
- ^ Courtwright, A.M.; He, X. (2002), “Dam1 is the Right One Phosphoregulation of Kinetochore Biorientation”, Developmental Cell 3 (5): 610–611, doi:10.1016/S1534-5807(02)00332-5, PMID 12431367
- ^ a b Cimini, D.; Moree, B.; Canman, J.C.; Salmon, E.D. (2003), “Merotelic kinetochore orientation occurs frequently during early mitosis in mammalian tissue cells and error correction is achieved by two different mechanisms”, Journal of Cell Science 116 (20): 4213–4225, doi:10.1242/jcs.00716, PMID 12953065
- ^ Adams, R.R.; Carmena, M.; Earnshaw, W.C. (2001), “Chromosomal passengers and the (aurora) ABCs of mitosis”, Trends in Cell Biology 11 (2): 49–54, doi:10.1016/S0962-8924(00)01880-8, PMID 11166196
- ^ Cheeseman, I.M.; Anderson, S.; Jwa, M.; Green, E.M.; Kang, J.; Yates, J.R.; Chan, C.S.M.; Drubin, D.G. et al. (2002), “Phospho-Regulation of Kinetochore-Microtubule Attachments by the Aurora Kinase Ipl1p”, Cell 111 (2): 163–172, doi:10.1016/S0092-8674(02)00973-X, PMID 12408861
- ^ Gautschi, Oliver; Heighway, Jim; Mack, Philip C.; Purnell, Phillip R.; Lara, Primo N.; Jr, .; Gandara, David R. (2008), “Aurora Kinases as Anticancer Drug Targets”, Clinical Cancer Research 14 (6): 1639–48, doi:10.1158/1078-0432.CCR-07-2179, PMID 18347165
- ^ Meraldi, Patrick; Draviam, Viji M.; Sorger, Peter K. (2004-07). “Timing and checkpoints in the regulation of mitotic progression”. Developmental Cell 7 (1): 45–60. doi:10.1016/j.devcel.2004.06.006. ISSN 1534-5807. PMID 15239953.
- ^ Tang, T.T.L.; Bickel, S.E.; Young, L.M.; Orr-weaver, T.L. (1998), “Maintenance of sister-chromatid cohesion at the centromere by the Drosophila MEI-S332 protein”, Genes & Development 12 (24): 3843–3856, doi:10.1101/gad.12.24.3843, PMC 317262, PMID 9869638
- ^ McGuinness, B.E.; Hirota, T.; Kudo, N.R.; Peters, J.M.; Nasmyth, K. (2005), “Shugoshin Prevents Dissociation of Cohesin from Centromeres During Mitosis in Vertebrate Cells”, PLOS Biol 3 (3): e86, doi:10.1371/journal.pbio.0030086, PMC 1054882, PMID 15737064
- ^ Joseph, Jomon; Tan, Shyh-Han; Karpova, Tatiana S.; McNally, James G.; Dasso, Mary (2002), “SUMO-1 targets RanGAP1 to kinetochores and mitotic spindles”, The Journal of Cell Biology 156 (4): 595–602, doi:10.1083/jcb.200110109, PMC 2174074, PMID 11854305
- ^ Arnaoutov, A.; Dasso, M. (2003), “The Ran GTPase Regulates Kinetochore Function”, Developmental Cell 5 (1): 99–111, doi:10.1016/S1534-5807(03)00194-1, PMID 12852855
- ^ Prasanth, S.G.; Prasanth, K.V.; Siddiqui, K.; Spector, D.L.; Stillman, B. (2004), “Human Orc2 localizes to centrosomes, centromeres and heterochromatin during chromosome inheritance”, The EMBO Journal 23 (13): 2651–2663, doi:10.1038/sj.emboj.7600255, PMC 449767, PMID 15215892
- ^ Shimada, K.; Gasser, S.M. (2007), “The Origin Recognition Complex Functions in Sister-Chromatid Cohesion in Saccharomyces cerevisiae”, Cell 128 (1): 85–99, doi:10.1016/j.cell.2006.11.045, PMID 17218257
- ^ Kato, H; Matsunaga, F; Miyazaki, S; Yin, L; D'urso, G; Tanaka, K; Murakami, Y (2008), “Schizosaccharomyces pombe Orc5 plays multiple roles in the maintenance of genome stability throughout the cell cycle”, Cell Cycle 7 (8): 1085–96, doi:10.4161/cc.7.8.5710, PMID 18414064
- ^ Skibbens, R.V.; Skeen, V.P.; Salmon, E.D. (1993), “Directional instability of kinetochore motility during chromosome congression and segregation in mitotic newt lung cells: a push-pull mechanism”, The Journal of Cell Biology 122 (4): 859–875, doi:10.1083/jcb.122.4.859, PMC 2119582, PMID 8349735
- ^ Rieder, C.L.; Salmon, E.D. (1994), “Motile kinetochores and polar ejection forces dictate chromosome position on the vertebrate mitotic spindle”, The Journal of Cell Biology 124 (3): 223–33, doi:10.1083/jcb.124.3.223, PMC 2119939, PMID 8294508
- ^ Skibbens, RV; Rieder, CL; Salmon, ED (1995), “Kinetochore motility after severing between sister centromeres using laser microsurgery: evidence that kinetochore directional instability and position is regulated by tension”, Journal of Cell Science 108 (7): 2537–48, PMID 7593295
- ^ Askham, J. M.; Vaughan, K. T.; Goodson, H. V.; Morrison, E. E. (2002), “Evidence That an Interaction between EB1 and p150Glued Is Required for the Formation and Maintenance of a Radial Microtubule Array Anchored at the Centrosome”, Molecular Biology of the Cell 13 (10): 3627–3645, doi:10.1091/mbc.E02-01-0061, PMC 129971, PMID 12388762
- ^ Schuyler, S.C.; Pellman, D. (2001), “Microtubule "Plus-End-Tracking Proteins" the End is Just the Beginning”, Cell 105 (4): 421–424, doi:10.1016/S0092-8674(01)00364-6, PMID 11371339
- ^ Howard, J.; Hyman, A.A. (2003), “Dynamics and mechanics of the microtubule plus end: cytoskeleton”, Nature 422 (6933): 753–758, doi:10.1038/nature01600, PMID 12700769
- ^ Green, R.A.; Wollman, R.; Kaplan, K.B. (2005), “APC and EB1 Function Together in Mitosis to Regulate Spindle Dynamics and Chromosome Alignment”, Molecular Biology of the Cell 16 (10): 4609–4622, doi:10.1091/mbc.E05-03-0259, PMC 1237068, PMID 16030254
- ^ Dujardin, D.; Wacker, U.I.; Moreau, A.; Schroer, T.A.; Rickard, J.E.; De Mey, J.R. (1998), “Evidence for a Role of CLIP-170 in the Establishment of Metaphase Chromosome Alignment”, The Journal of Cell Biology 141 (4): 849–862, doi:10.1083/jcb.141.4.849, PMC 2132766, PMID 9585405
- ^ Maiato, H.; Khodjakov, A.; Rieder, C.L. (2004), “Drosophila CLASP is required for the incorporation of microtubule subunits into fluxing kinetochore fibres”, Nature Cell Biology 7 (1): 42–47, doi:10.1038/ncb1207, PMC 2596653, PMID 15592460
- ^ Maiato, H.; Fairley, E.A.L.; Rieder, C.L.; Swedlow, J.R.; Sunkel, C.E.; Earnshaw, W.C. (2003), “Human CLASP1 is an Outer Kinetochore Component that Regulates Spindle Microtubule Dynamics”, Cell 113 (7): 891–904, doi:10.1016/S0092-8674(03)00465-3, hdl:10216/53832, PMID 12837247
関連項目
[編集]外部リンク
[編集]Text is available under the CC BY-SA 4.0 license; additional terms may apply.
Images, videos and audio are available under their respective licenses.
