エキソソーム複合体

エキソソーム複合体(エキソソームふくごうたい、英: Exosome Complex、PM/Scl複合体、単にエキソソーム、エクソソームとも)は、真核生物、古細菌の核に存在し、様々なRNAを分解する多タンパク質複合体である。細菌ではデグラドソームと呼ばれる複合体が似た役割を果たす。
エキソソームの中心部は他のタンパク質が付随した6つのタンパク質から構成されている。真核細胞では、エキソソームは細胞全体に存在するが、特に核小体に多い。だが、複合体を制御するタンパク質が異なるため、存在する場所によって活性、基質特異性が異なる。エキソソームの基質はmRNA、rRNA、ncRNA等である。エキソソームはエキソリボヌクレアーゼ活性を有し、RNAを3'末端から分解する。また、真核細胞の場合はエンドリボヌクレアーゼ活性も有し、RNA鎖の途中を切断することもできる。
エキソソームを直接の原因とする疾患は知られていないが、いくつかの自己免疫疾患(特に筋炎/強皮症重複症候群)では、エキソソームの構成タンパク質を標的とする自己抗体が生産される。エキソソームを阻害することで効果を示すがん化学療法剤もある。
発見
[編集]1997年、エキソソームは出芽酵母からリボヌクレアーゼの一種として発見された[1]。だが1999年、それはヒト細胞で自己免疫疾患の抗原として特定されていたPM/Scl複合体と相同であることが明らかになった(#疾患を参照)[2]。この「PM/Scl複合体」を精製することで、その構成タンパク質を全て特定することができた[3][4]。古細菌では、2001年になって利用可能なゲノムデータが増え、その2年前に精製されていたタンパク質がエキソソーム複合体だと予測された[5][6]。
構造
[編集]中心タンパク
[編集]
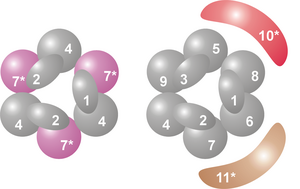
複合体の中心部では、6個のRNase PH様リボヌクレアーゼが環構造を形成している[7]。古細菌では2種のユニッ(Rrp41、Rrp42と呼ばれる)3個ずつで構成されるが、 真核細胞では6種の異なったユニットで構成される[8][9]。6種のユニットの内、3種はRrp41に、残り3種はRrp42に類似している[10]。
この環構造の上部には、S1RNA結合ドメインを持ったユニットが3個乗っている。その内2個のユニットはK-homology(KH)ドメインも有する[7]。真核細胞では3つのタンパク質は全て異なるが、古細菌ではその内2つが同じタンパク質である[11]。

この環構造はRNase PHやPNPaseによく似ている。細菌では、RNase PHユニットが6量化、環構造を形成しtRNAの転写後修飾を行っている[12]。細菌、葉緑体、ミトコンドリアで見られるRNA加リン酸分解酵素PNPaseでは、S1、KH、2つのRNase PHドメインを持ったタンパクが3量体を形成し、エキソソームとほぼ同一の複合体となっている[13][14]。構成タンパクのドメイン、構造の類似性から、これらの複合体には進化的関連があり、共通祖先を持つと考えられている[15]。RNase PH様エキソソームタンパク、PNPase、RNase PHは全てRNase PHファミリーに属し、酵素活性がある場合には加リン酸分解を行う[7]。
付随タンパク
[編集]真核細胞ではこれらの9個のユニットに加え、さらに2個のユニットが付随することが多い。その内の一つがRNase Rファミリーに属する加水分解リボヌクレアーゼRrp44である。Rrp44はエキソリボヌクレアーゼ、エンドリボヌクレアーゼ双方の活性を持つが、その機能はRrp44内の別々のドメインが担当する[16][17]。酵母では、Rrp44は全てのエキソソーム複合体に付随しており、その活性に重要である[18]。ヒト細胞にもこのタンパク質ホモログが存在するにもかかわらず、長い間それがヒトエキソソーム複合体に付随するという証拠は得られなかった[7]。2010年になって、ヒトのRrp44ホモログが3つ発見され、そのうち2つが複合体に付随することが分かった。その内1つ (Dis3L1) は細胞質に、1つ (Dis3) は核に局在するため、 異なったRNA基質を分解する[19][20]。

2つ目のタンパクは、酵母ではRrp6、ヒトではPM/Scl-100とよばれる。Rrp44と同じく加水分解性エキソリボヌクレアーゼだが、このタンパクはRNase Dファミリーに属する[21]。PM/Scl-100は核エキソソームの一部だが、細胞質エキソソームの構成要素とも成り得る[22]。
制御タンパク
[編集]付随タンパクとは別に、エキソソーム複合体と相互作用する多くのタンパクが存在する。これらの緩く結合したタンパクは、複合体の活性と基質特異性を制御すると考えられている。細胞質では、エキソソームはmRNAの分解を制御するAUリッチ領域 (ARE) 結合タンパク(例えばw:KHSRP、TTP)と相互作用する。核ではRNA結合タンパク(例えばMPP6/Mpp6、C1D/Rrp47 それぞれヒト/酵母)が特定の基質を処理するために必要である[7]。
エキソソームと相互作用するタンパク質複合体も存在する。細胞質では、RNAヘリカーゼ (Ski2) を含み、mRNAをエキソソームまで誘導するSki複合体がある[23]。核では、エキソソームによるrRNA、snoRNAの転写後修飾を、RNAヘリカーゼ (Mtr4)、ポリアデニル化 (Trf4) 活性を持つTRAMP複合体が調節している[24]。
機能
[編集]酵素
[編集]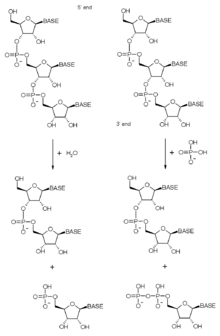
既に示したように、エキソソームはリボヌクレアーゼ活性を持つタンパク質を多く含む。エキソソームは元々、RNA分子を3'末端から分解する3'-5'エキソリボヌクレアーゼだったが、これらのドメインは、進化にともなって様々な活性を得たり、失ったりしてきた。エキソソームに含まれるエキソリボヌクレアーゼは加リン酸分解(RNase PH様タンパク)か、真核細胞では加水分解(RNase R、RNase Dドメインタンパク)酵素である。加リン酸分解酵素はホスホジエステル結合の切断に無機リン酸を用い、ヌクレオチド二リン酸を放出する。加水分解酵素は切断に水を用い、ヌクレオチド一リン酸を放出する。
古細菌では、Rrp41サブユニットは加リン酸分解エキソリボヌクレアーゼである。このユニットが環構造内に3個存在し、酵素活性を担う[9]。真核細胞では、RNase PHサブユニットには酵素活性はない。つまり、環構造内に酵素活性のあるタンパクは含まれていない[25]。それにも関わらず、エキソソーム中心部の構造は古細菌からヒトまで高度に保存されており、何らかの重要な細胞機能を持つと考えられている。真核細胞では加リン酸分解活性が欠如する代わりに、別の加水分解酵素がリボヌクレアーゼ活性を担う[26][27][28]。
酵母のエキソソームには加水分解酵素Rrp6、Rrp44が付随しているが、ヒトではRrp44の位置にDis3またはDis3L1が入っている[19][20]。元々、S1ドメインを持つタンパクが3'-5'加水分解性エキソリボヌクレアーゼ活性を持つと考えられていたが、最近ではその活性の存在は疑問視されており、分解前の基質を複合体に結合しておく役割を持つと考えられている[26]。
基質
[編集]エキソソームは様々なRNAの分解、転写後修飾に関わっている。細胞質ではmRNAの代謝回転に関わり、ナンセンス仲介減衰またはノンストップ分解経路によってエラータグが付けられたmRNAを分解できる。mRNAは通常の代謝回転によっても分解される。また、mRNAの3' 非翻訳領域に存在するAUリッチ領域への結合を介してその安定性を制御するいくつかのタンパクがエキソソームと相互作用する[29][30][31]。細胞核では、エキソソームは核内低分子RNAの正しい処理に必要である[32]。核小体はエキソソームの大部分が検出された区画であり、5.8SrRNAの修飾(最初に特定されたエキソソームの機能)やsnoRNAの修飾を行う[1][32][33]。
ほとんどの細胞はエキソソーム複合体の他にも3'末端または5'末端からRNAを分解する酵素を持つが、エキソソームは細胞の生存に不可欠である。RNA干渉等でエキソソーム構成タンパクの発現量を減らすと、細胞の成長は停止し、遂には死ぬ。中心タンパクと2つの付随タンパクは同じくらい重要である[34]。細菌はエキソソームを持たないが、似た機能とより簡単な構造を持ち、PNPaseを含む「デグラドソーム」という構造を持つ[35]。

エキソソームはRNAの品質管理に重要である。原核生物とは違い、真核生物は高活性のRNA監視機構を持つ。この機構はリボソーム等が作られる際に、タンパク質と未修飾、誤修飾されたRNAとの複合体が核から出ることを防ぐ。つまり、タンパク質合成等の重要なプロセスに異常な複合体が関わることを妨げる役割を持つと推定されている[36]。
エキソソームは修飾、代謝回転、監視の役割に加え、酵母ゲノム内の多くの遺伝子座から生産されるCUTsと呼ばれる転写産物の分解に重要である[37][38]。この不安定なRNAとその分解機構は明らかになっていないが、ヒト細胞からも似たRNAが発見されている[39]
疾患
[編集]自己免疫
[編集]エキソソーム複合体は様々な自己免疫疾患で自己抗体の標的となる。この自己抗体は、強皮症に多発筋炎又は皮膚筋炎が合併した症状を示す自己免疫疾患、筋炎/強皮症重複症候群の患者に見られる。[40]。自己抗体は患者の血清から検出される。過去には、仔牛胸腺抽出物を用いた二重免疫拡散法、HEp-2細胞(ヒト喉頭癌由来培養細胞)上での免疫蛍光染色、ヒト細胞抽出物を用いた免疫沈降法などで診断されていた。エキソソーム抗体陽性血清を用いた免疫沈降法では、特徴的なタンパク質群が沈殿する。エキソソーム複合体が同定される前に、既にこのパターンはPM/Scl複合体と呼ばれていた[41]。患者の血清を免疫蛍光染色すると核小体が染色されるため、自己抗体の抗原はリボソーム生合成に関わっていると推測された[42]。最近では組み換えエキソソームタンパク質が利用可能になり、抗体を検出するラインイムノアッセイ (LIA) やELISAに利用されている[7]。
これらの疾患では、抗体はPM/Scl-100(RNase D様タンパク質)、PM/Scl-75(環構造内のRNase PH様タンパク質)を主な標的としており、PM/Scl重複症候群患者の約30%でそのような抗体が見られる[43]。また、他のサブユニットや付随タンパク質(特にC1D)も標的と成り得る[44][45]。現在最も感度の高い方法は、ELISAの抗原としてPM/Scl-100タンパク質由来のペプチドを使うものである。この方法を用いると、PM/Scl重複症候群患者の55%以上から自己抗体が検出されるが、強皮症、多発筋炎、皮膚筋炎等を単独で罹患している患者からも抗体が検出される[46]。
自己抗体は複数の異なる自己免疫疾患の患者から検出されるため、その臨床症状は多岐に渡る。最もよく見られる症状は、レイノー病、関節炎、筋炎、強皮症等の個々の自己免疫疾患に典型的な症状である[47]。これらの患者の治療は対症療法的で、個々の自己免疫疾患に用いられる免疫抑制剤、調節薬などを用いることになる[48]。
癌治療
[編集]エキソソームは癌化学療法に用いられるフルオロウラシルで阻害される。これは固形癌に対する最も成功した治療薬である。フルオロウラシルで処理した酵母は、分子生物学的方法でエキソソームを阻害した時と同じく、rRNAの修飾に異常をきたす。rRNAの正しい修飾を欠くことは細胞にとって致命的であり、それはこの薬の代謝拮抗作用を説明する[49]。
サブユニット一覧
[編集]| Legend | General name | Domains | Human | Yeast (S. cerevisiae) | Archaea | MW (kD) | Human gene | Yeast gene |
|---|---|---|---|---|---|---|---|---|
| 1 | Csl4 | S1 RBD | hCsl4 | Csl4p/Ski4p | Csl4 | 21-32 | EXOSC1 | YNL232W |
| 2 | Rrp4 | S1/KH RBD | hRrp4 | Rrp4p | Rrp4 | 28-39 | EXOSC2 | YHR069C |
| 3 | Rrp40 | S1/KH RBD | hRrp40 | Rrp40p | (Rrp4)A | 27-32 | EXOSC3 | YOL142W |
| 4 | Rrp41 | RNase PH | hRrp41 | Rrp41p/Ski6p | Rrp41C | 26-28 | EXOSC4 | YGR195W |
| 5 | Rrp46 | RNase PH | hRrp46 | Rrp46p | (Rrp41)A,C | 25-28 | EXOSC5 | YGR095C |
| 6 | Mtr3 | RNase PH | hMtr3 | Mtr3p | (Rrp41)A,C | 24-37 | EXOSC6 | YGR158C |
| 7 | Rrp42 | RNase PH | hRrp42 | Rrp42p | Rrp42 | 29-32 | EXOSC7 | YDL111C |
| 8 | Rrp43 | RNase PH | OIP2 | Rrp43p | (Rrp42)A | 30-44 | EXOSC8 | YCR035C |
| 9 | Rrp45 | RNase PH | PM/Scl-75 | Rrp45p | (Rrp42)A | 34-49 | EXOSC9 | YDR280W |
| 10 | Rrp6 | RNase D | PM/Scl-100C | Rrp6pC | n/a | 84-100 | EXOSC10 | YOR001W |
| 11 | Rrp44 | RNase R | Dis3B,C | Rrp44p/Dis3pC | n/a | 105-113 | DIS3
DIS3L1 |
YOL021C |
- A 古細菌の複合体中心部のいくつかのユニットは、同じタンパク質のコピーである。
- B ヒトでは2種類のタンパク質がこの位置に入ることができる。細胞質ではDis3L1、核ではDis3が入る。
- C 複合体のリボヌクレアーゼ活性を担う。
出典
[編集]- ^ a b Mitchell, P; Petfalski, E; Shevchenko, A; Mann, M; Tollervey, D (1997), “The Exosome: A Conserved Eukaryotic RNA Processing Complex Containing Multiple 3′→5′ Exoribonucleases”, Cell 91 (4): 457–466, doi:10.1016/S0092-8674(00)80432-8, PMID 9390555
- ^ Allmang, C; Petfalski, E; Podtelejnikov, A; Mann, M; Tollervey, D; Mitchell, P (1999), “The yeast exosome and human PM-Scl are related complexes of 3' --> 5' exonucleases”, Genes and Development 13 (16): 2148–58, doi:10.1101/gad.13.16.2148, PMC 316947, PMID 10465791
- ^ Brouwer, R; Allmang, C; Raijmakers, R; Van Aarssen, Y; Egberts, WV; Petfalski, E; Van Venrooij, WJ; Tollervey, D et al. (2001), “Three novel components of the human exosome”, Journal of Biological Chemistry 276 (9): 6177–84, doi:10.1074/jbc.M007603200, PMID 11110791
- ^ Chen, CY; Gherzi, R; Ong, SE; Chan, EL; Raijmakers, R; Pruijn, GJ; Stoecklin, G; Moroni, C et al. (2001), “AU binding proteins recruit the exosome to degrade ARE-containing mRNAs”, Cell 107 (4): 451–64, doi:10.1016/S0092-8674(01)00578-5, PMID 11719186
- ^ Koonin, EV; Wolf, YI; Aravind, L (2001), “Prediction of the archaeal exosome and its connections with the proteasome and the translation and transcription machineries by a comparative-genomic approach”, Genome Research 11 (2): 240–52, doi:10.1101/gr.162001, PMC 311015, PMID 11157787
- ^ Evguenieva-Hackenberg, E; Walter, P; Hochleitner, E; Lottspeich, F; Klug, G (2003), “An exosome-like complex in Sulfolobus solfataricus”, EMBO Reports 4 (9): 889–93, doi:10.1038/sj.embor.embor929, PMC 1326366, PMID 12947419
- ^ a b c d e f Schilders, G; Van Dijk, E; Raijmakers, R; Pruijn, GJ (2006), “Cell and molecular biology of the exosome: how to make or break an RNA”, International review of cytology 251: 159–208, doi:10.1016/S0074-7696(06)51005-8, PMID 16939780
- ^ Lorentzen, E; Walter, P; Fribourg, S; Evguenieva-Hackenberg, E; Klug, G; Conti, E (2005), “The archaeal exosome core is a hexameric ring structure with three catalytic subunits”, Nature Structural & Molecular Biology 12 (7): 575–81, doi:10.1038/nsmb952, PMID 15951817
- ^ a b Shen, V; Kiledjian, M (2006), “A view to a kill: structure of the RNA exosome”, Cell 127 (6): 1093–5, doi:10.1016/j.cell.2006.11.035, PMC 1986773, PMID 17174886
- ^ Raijmakers, R; Egberts, WV; Van Venrooij, WJ; Pruijn, GJ (2002), “Protein-protein interactions between human exosome components support the assembly of RNase PH-type subunits into a six-membered PNPase-like ring”, Journal of Molecular Biology 323 (4): 653–63, doi:10.1016/S0022-2836(02)00947-6, PMID 12419256
- ^ Walter, P; Klein, F; Lorentzen, E; Ilchmann, A; Klug, G; Evguenieva-Hackenberg, E (2006), “Characterization of native and reconstituted exosome complexes from the hyperthermophilic archaeon Sulfolobus solfataricus”, Molecular Microbiology 62 (4): 1076–89, doi:10.1111/j.1365-2958.2006.05393.x, PMID 17078816
- ^ Ishii, R; Nureki, O; Yokoyama, S (2003), “Crystal structure of the tRNA processing enzyme RNase PH from Aquifex aeolicus”, Journal of Biological Chemistry 278 (34): 32397–404, doi:10.1074/jbc.M300639200, PMID 12746447
- ^ Symmons, MF; Jones, GH; Luisi, BF (2000), “A duplicated fold is the structural basis for polynucleotide phosphorylase catalytic activity, processivity, and regulation”, Structure 8 (11): 1215–26, doi:10.1016/S0969-2126(00)00521-9, PMID 11080643
- ^ Harlow, LS; Kadziola, A; Jensen, KF; Larsen, S (2004), “Crystal structure of the phosphorolytic exoribonuclease RNase PH from Bacillus subtilis and implications for its quaternary structure and tRNA binding”, Protein Science 13 (3): 668–77, doi:10.1110/ps.03477004, PMC 2286726, PMID 14767080
- ^ Lin-Chao, S; Chiou, NT; Schuster, G (2007), “The PNPase, exosome and RNA helicases as the building components of evolutionarily-conserved RNA degradation machines”, Journal of Biomedical Science 14 (4): 523–32, doi:10.1007/s11373-007-9178-y, PMID 17514363
- ^ Lebreton, A; Tomecki, R; Dziembowski, A; Séraphin, B (2008), “Endonucleolytic RNA cleavage by a eukaryotic exosome.”, Nature 456 (7224): 993–6, doi:10.1038/nature07480, PMID 19060886
- ^ Schneider, C; Leung, E; Brown, J; Tollervey, D (2009), “The N-terminal PIN domain of the exosome subunit Rrp44 harbors endonuclease activity and tethers Rrp44 to the yeast core exosome.”, Nucleic Acids Research 37 (4): 1127–40, doi:10.1093/nar/gkn1020, PMC 2651783, PMID 19129231
- ^ Schneider, C; Anderson, JT; Tollervey, D (2007), “The exosome subunit Rrp44 plays a direct role in RNA substrate recognition”, Molecular Cell 27 (2): 324–31, doi:10.1016/j.molcel.2007.06.006, PMID 17643380
- ^ a b Staals, RH; Bronkhorst, AW; Schilders, G; Slomovic, S; Schuster, G; Heck, AJ; Raijmakers, R; Pruijn, GJ (2010), “Dis3-like 1: a novel exoribonuclease associated with the human exosome.”, The EMBO journal 29 (14): 2358–67, doi:10.1038/emboj.2010.122, PMC 2910272, PMID 20531389
- ^ a b Tomecki, R; Kristiansen, MS; Lykke-Andersen, S; Chlebowski, A; Larsen, KM; Szczesny, RJ; Drazkowska, K; Pastula, A et al. (2010), “The human core exosome interacts with differentially localized processive RNases: hDIS3 and hDIS3L.”, The EMBO journal 29 (14): 2342–57, doi:10.1038/emboj.2010.121, PMC 2910271, PMID 20531386
- ^ Mian, IS (1997), “Comparative sequence analysis of ribonucleases HII, III, II PH and D”, Nucleic Acids Research 25 (16): 3187–3195, doi:10.1093/nar/25.16.3187, PMC 146874, PMID 9241229
- ^ Raijmakers, R; Schilders, G; Pruijn, GJ (2004), “The exosome, a molecular machine for controlled RNA degradation in both nucleus and cytoplasm”, European Journal of Cell Biology 83 (5): 175–83, doi:10.1078/0171-9335-00385, PMID 15346807
- ^ Wang, L; Lewis, MS; Johnson, AW (2005), “Domain interactions within the Ski2/3/8 complex and between the Ski complex and Ski7p”, RNA 11 (8): 1291–302, doi:10.1261/rna.2060405, PMC 1370812, PMID 16043509
- ^ LaCava, J; Houseley, J; Saveanu, C; Petfalski, E; Thompson, E; Jacquier, A; Tollervey, D (2005), “RNA degradation by the exosome is promoted by a nuclear polyadenylation complex”, Cell 121 (5): 713–24, doi:10.1016/j.cell.2005.04.029, PMID 15935758
- ^ Liu, Q; Greimann, JC; Lima, CD (2007), “Erratum: Reconstitution, activities, and structure of the eukaryotic RNA exosome”, Cell 131 (1): 188–189, doi:10.1016/j.cell.2007.09.019
- ^ a b Dziembowski, A; Lorentzen, E; Conti, E; Séraphin, B (2007), “A single subunit, Dis3, is in essence responsible for yeast exosome core activity”, Nature Structural & Molecular Biology 14 (1): 15–22, doi:10.1038/nsmb1184, PMID 17173052
- ^ Liu, Q; Greimann, JC; Lima, CD (2006), “Reconstitution, activities, and structure of the eukaryotic RNA exosome”, Cell 127 (6): 1223–37, doi:10.1016/j.cell.2006.10.037, PMID 17174896
- ^ Lorentzen, E; Conti, E (2005), “Structural basis of 3' end RNA recognition and exoribonucleolytic cleavage by an exosome RNase PH core”, Molecular Cell 20 (3): 473–81, doi:10.1016/j.molcel.2005.10.020, PMID 16285928
- ^ LeJeune, F; Li, X; Maquat, LE (2003), “Nonsense-mediated mRNA decay in mammalian cells involves decapping, deadenylating, and exonucleolytic activities”, Molecular Cell 12 (3): 675–87, doi:10.1016/S1097-2765(03)00349-6, PMID 14527413
- ^ Wilson, MA; Meaux, S; Van Hoof, A (2007), “A genomic screen in yeast reveals novel aspects of nonstop mRNA metabolism”, Genetics 177 (2): 773–84, doi:10.1534/genetics.107.073205, PMC 2034642, PMID 17660569
- ^ Lin, WJ; Duffy, A; Chen, CY (2007), “Localization of AU-rich element-containing mRNA in cytoplasmic granules containing exosome subunits”, Journal of Biological Chemistry 282 (27): 19958–68, doi:10.1074/jbc.M702281200, PMID 17470429
- ^ a b Allmang, C; Kufel, J; Chanfreau, G; Mitchell, P; Petfalski, E; Tollervey, D (1999), “Functions of the exosome in rRNA, snoRNA and snRNA synthesis”, EMBO Journal 18 (19): 5399–410, doi:10.1093/emboj/18.19.5399, PMC 1171609, PMID 10508172
- ^ Schilders, G; Raijmakers, R; Raats, JM; Pruijn, GJ (2005), “MPP6 is an exosome-associated RNA-binding protein involved in 5.8S rRNA maturation”, Nucleic Acids Research 33 (21): 6795–804, doi:10.1093/nar/gki982, PMC 1310903, PMID 16396833
- ^ van Dijk, EL; Schilders, G; Pruijn, GJ (2007), “Human cell growth requires a functional cytoplasmic exosome, which is involved in various mRNA decay pathways”, RNA 13 (7): 1027–35, doi:10.1261/rna.575107, PMC 1894934, PMID 17545563
- ^ Carpousis AJ, AJ (2002), “The Escherichia coli RNA degradosome: structure, function and relationship in other ribonucleolytic multienzyme complexes”, Biochem. Soc. Trans. 30 (2): 150–5, doi:10.1042/BST0300150, PMID 12035760
- ^ Houseley J, LaCava J, Tollervey D, J; Lacava, J; Tollervey, D (July 2006), “RNA-quality control by the exosome”, Nat. Rev. Mol. Cell Biol. 7 (7): 529–39, doi:10.1038/nrm1964, PMID 16829983
- ^ Wyers F, Rougemaille M, Badis G, et al, F; Rougemaille, M; Badis, G; Rousselle, JC; Dufour, ME; Boulay, J; Régnault, B; Devaux, F et al. (June 2005), “Cryptic pol II transcripts are degraded by a nuclear quality control pathway involving a new poly(A) polymerase”, Cell 121 (5): 725–37, doi:10.1016/j.cell.2005.04.030, PMID 15935759
- ^ Neil H, Malabat C, d'Aubenton-Carafa Y, Xu Z, Steinmetz LM, Jacquier A, H; Malabat, C; D'aubenton-Carafa, Y; Xu, Z; Steinmetz, LM; Jacquier, A (February 2009), “Widespread bidirectional promoters are the major source of cryptic transcripts in yeast”, Nature 457 (7232): 1038–42, doi:10.1038/nature07747, PMID 19169244
- ^ Preker P, Nielsen J, Kammler S, et al, P; Nielsen, J; Kammler, S; Lykke-Andersen, S; Christensen, MS; Mapendano, CK; Schierup, MH; Jensen, TH (December 2008), “RNA exosome depletion reveals transcription upstream of active human promoters”, Science 322 (5909): 1851–4, doi:10.1126/science.1164096, PMID 19056938
- ^ J.E. Pope, JE (2002), “Scleroderma overlap syndromes”, Current Opinion in Rheumatology 14 (6): 704–10, doi:10.1097/00002281-200211000-00013, PMID 12410095
- ^ Gelpi, C; Algueró, A; Angeles Martinez, M; Vidal, S; Juarez, C; Rodriguez-Sanchez, JL (1991), “Identification of protein components reactive with anti-PM/Scl autoantibodies”, Clinical and Experimental Immunology 81 (1): 59–64, doi:10.1111/j.1365-2249.1990.tb05291.x, PMC 1535032, PMID 2199097
- ^ Targoff, IN; Reichlin, M (1985), “Nucleolar localization of the PM-Scl antigen”, Arthritis & Rheumatism 28 (2): 226–30, doi:10.1002/art.1780280221, PMID 3918546
- ^ Raijmakers, R; Renz, M; Wiemann, C; Egberts, WV; Seelig, HP; Van Venrooij, WJ; Pruijn, GJ (2004), “PM-Scl-75 is the main autoantigen in patients with the polymyositis/scleroderma overlap syndrome”, Arthritis & Rheumatism 50 (2): 565–9, doi:10.1002/art.20056, PMID 14872500
- ^ Brouwer, R; Vree Egberts, WT; Hengstman, GJ; Raijmakers, R; Van Engelen, BG; Seelig, HP; Renz, M; Mierau, R et al. (2002), “Autoantibodies directed to novel components of the PM/Scl complex, the human exosome.”, Arthritis Research 4 (2): 134–8, doi:10.1186/ar389, PMC 83843, PMID 11879549
- ^ Schilders, G; Egberts, WV; Raijmakers, R; Pruijn, GJ (2007), “C1D is a major autoantibody target in patients with the polymyositis-scleroderma overlap syndrome”, Arthritis & Rheumatism 56 (7): 2449–54, doi:10.1002/art.22710, PMID 17599775
- ^ Mahler, M; Raijmakers, R; Dähnrich, C; Blüthner, M; Fritzler, MJ (2005), “Clinical evaluation of autoantibodies to a novel PM/Scl peptide antigen”, Arthritis Research & Therapy 7 (3): R704–13, doi:10.1186/ar1729, PMC 1174964, PMID 15899056
- ^ Mahler, M; Raijmakers, R (2007), “Novel aspects of autoantibodies to the PM/Scl complex: Clinical, genetic and diagnostic insights”, Autoimmunity Reviews 6 (7): 432–7, doi:10.1016/j.autrev.2007.01.013, PMID 17643929
- ^ Jablonska, S; Blaszczyk, M (1998), “Scleromyositis: a scleroderma/polymyositis overlap syndrome”, Clinical Rheumatology 17 (6): 465–7, doi:10.1007/BF01451281, PMID 9890673
- ^ Lum, PY; Armour, CD; Stepaniants, SB; Cavet, G; Wolf, MK; Butler, JS; Hinshaw, JC; Garnier, P et al. (2004), “Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes”, Cell 116 (1): 121–37, doi:10.1016/S0092-8674(03)01035-3, PMID 14718172
参考文献
[編集]- Schilders, G; Pruijn, GJ (2008), “Biochemical studies of the mammalian exosome with intact cells”, Methods Enzymol 448: 211–226, doi:10.1016/S0076-6879(08)02611-6, PMID 19111178
- Houseley, J; Tollervey, D (2008), “The nuclear RNA surveillance machinery: the link between ncRNAs and genome structure in budding yeast?”, Biochim Biophys Acta 1779 (4): 239–246, doi:10.1016/j.bbagrm.2007.12.008, PMID 18211833
- Vanacova, S; Stefl, R (2007), “The exosome and RNA quality control in the nucleus”, EMBO reports 8 (7): 651–657, doi:10.1038/sj.embor.7401005, PMC 1905902, PMID 17603538
- Houseley, J; Lacava, J; Tollervey, D (2006), “RNA-quality control by the exosome”, Nature Reviews Molecular Cell Biology 7 (7): 529–539, doi:10.1038/nrm1964, PMID 16829983 –-- subscription required
- Büttner, K; Wenig, K; Hopfner, KP (2006), “The exosome: a macromolecular cage for controlled RNA degradation”, Molecular Microbiology 61 (6): 1372–1379, doi:10.1111/j.1365-2958.2006.05331.x, PMID 16968219
- Lorentzen, E; Conti, E (2006), “The Exosome and the Proteasome: Nano-Compartments for Degradation”, Cell 125 (4): 651–654, doi:10.1016/j.cell.2006.05.002, PMID 16713559
- Pruijn, GJ (2005), “Doughnuts dealing with RNA”, Nature Structural & Molecular Biology 12 (7): 562–564, doi:10.1038/nsmb0705-562, PMID 15999107
関連項目
[編集]外部リンク
[編集]Text is available under the CC BY-SA 4.0 license; additional terms may apply.
Images, videos and audio are available under their respective licenses.
