ポリアデニル化

ポリアデニル化(ポリアデニルか、英語: Polyadenylation)はRNAにポリA鎖(poly-A tail)を付加することである。ポリA鎖は多数のAMPから構成されており、RNAをアデニン塩基で伸長することに相当する。真核生物では、ポリアデニル化は翻訳可能な成熟mRNAを生産するために不可欠であり、広い意味では遺伝子発現過程の一部であるといえる。
ポリアデニル化は転写終了時から始まる。特定のタンパク質複合体がRNA3' 末端のセグメントを切り離し、そこからポリA鎖を合成する。いくつかの遺伝子では、切断できる部位が複数あり、その内1箇所にポリA鎖が追加される。そのため、ポリアデニル化は選択的スプライシングのように、1つの遺伝子から複数の転写産物を作り出す[1]。
ポリA鎖はmRNAの安定性に関わり、核外輸送、翻訳に重要である。これは時間と共に短くなり、十分に短くなった時点でmRNAは酵素により分解される[2]。だが、少数の細胞では、ポリA鎖の短いmRNAが再度のポリアデニル化に備えて細胞質に蓄えられている[3]。細菌ではこれと反対に、ポリアデニル化はRNAの分解を引き起こす[4]。これは真核細胞の非コードRNAでも見られる[5]。ポリアデニル化が生物全般に見られることは、これが生命の歴史の中で早い段階に進化したことを意味する。
背景
[編集]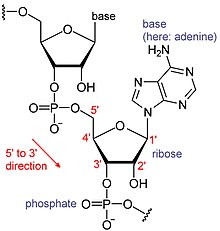
RNAは巨大な生体分子で、ヌクレオチドが繋がって構成されている。ポリA(ポリアデニル酸)[6]とは、RNA塩基(A:アデニン・U:ウラシル・G:グアニン・C:シトシン)のうち、Aのみが繋がったものである。RNAは通常、鋳型DNAから転写されることで合成される。転写は5' 末端から3' 末端に向けて進むため、塩基配列も5'から3'に向けて表記される。ポリA鎖が付加されるのは3' 末端である[1][7]。
伝令RNA(mRNA)は翻訳されてタンパク合成の鋳型となるコーディング領域を含む。その他の部分は非翻訳領域と呼ばれ、mRNAの活性を制御している[8]。非コードRNAという翻訳されないRNAも多くあり、非翻訳領域と同じように様々な制御を行う[9]。
核ポリアデニル化
[編集]機能
[編集]核において、ポリA化は転写終了時に行われる。ポリA鎖はmRNAを細胞質での分解から保護し、転写終結、核外輸送、翻訳を補助する[2]。真核細胞のmRNAはほぼ全てポリA化されているが[10]、動物の複製依存的ヒストンmRNAは例外である[11]。これは真核細胞のmRNAがポリA鎖を欠く唯一の例で、プリンリッチ配列に続くステムループ構造で終端し、切断箇所を示している。この構造は"ヒストン下流要素"(HDE)と呼ばれる[12]。
多くの真核細胞の非コードRNAは転写終了時にポリA化される。miRNAのように、末端がプロセシングで除かれるため、転写中間体にはポリA鎖があるが成熟RNAにはないものもある[13][14]。だが例えば、X染色体の不活性化を調節するXistなど、遺伝子発現制御を行う多くの長鎖非コードRNAでは、ポリA鎖は成熟RNAの一部である[15]。
機構
[編集]| 構成要素:[10] CPSF: 切断・ポリアデニル化因子 |
真核細胞核でのポリアデニル化の対象は、RNAポリメラーゼIIにより作られるmRNA前駆体などである。多タンパク複合体(構成要素は右表)が3' 末端近くを開裂し、そこからポリA化が始まる。開裂は酵素CPSFによって触媒され[11]、結合部位から10–30塩基下流で起こる[16]。この部位にはよくAAUAAA配列があるが、多少変化してもCPSFは結合することができる[17]。別の2つのタンパク、CstFとCFIもこれに関与し、CstFはCPSF結合部位の下流にあるGUリッチ領域と結合する[18]。CFIはさらに別の箇所(哺乳類ではUGUAA配列[19][20][21])を認識し、AAUAAA配列が失われてもCPSFを媒介することができる[22][23]。ポリアデニル化シグナル(開裂複合体に認識される配列モチーフ)は分類群によって変化する。ヒトのポリA化部位はほぼAAUAAA配列を含むが[18]、この配列は植物・菌類では珍しい[24]。
CstFはRNAポリメラーゼIIとも結合しているため、転写後すぐに開裂を行うことができる[25]。詳しくは分かっていないが、開裂にはCFIIというタンパクも関わる[26]。ポリA化シグナルに伴う開裂部位は、50塩基程度は変化することができる[27]。
RNAが開裂するとポリAポリメラーゼによるポリA化が始まり、ATPを用いてポリA鎖を伸長させていく[28]。その後、別のタンパクPAB2が短く新しいポリA鎖に結合し、RNAとポリAポリメラーゼの親和性を増加させる。ポリA鎖が約250塩基に達すると酵素はCPSFから外れ、ポリA化は終了する[29][30]。CPSFはRNAポリメラーゼIIと接触し、転写終結を伝達する[31][32]。ポリA化機構はRNAイントロンを除去するスプライソソームとも物理的に結合している[23]。
下流作用
[編集]ポリA鎖はポリA結合タンパク質結合部位として機能する。ポリA結合タンパクはRNAの核外移送・翻訳を促進し、分解を妨げる[33]。酵母では、ポリA鎖を短縮しmRNAの核外移送を可能にするポリAヌクレアーゼの結合も媒介する。mRNAはポリA結合タンパクと共に細胞質に移送されるが、移送されなかったmRNAはエキソソーム複合体によって分解される[34][35]。ポリA結合タンパクは翻訳に影響する様々なタンパクの結合も媒介し[34]、その内の1つが40Sリボソームサブユニットを媒介するeIF4Gである[36]。だが、ポリA鎖は全てのmRNAの翻訳に不可欠というわけではない[37]。
脱アデニル化
[編集]真核生物体細胞では、細胞質にあるmRNAのポリA鎖は次第に短くなり、翻訳が妨げられて分解が促進される[38]。だが、これが起こるにはかなり時間がかかる[39]。このプロセスは、mRNAの3' 非翻訳領域と相補的なmiRNAによって加速される[40]。卵母細胞では、ポリA鎖が短縮されたmRNAは分解されないが、代わりに翻訳されずに貯蔵される。このRNAは卵胞形成中に、細胞質でのポリA化によって再活性化される[41]。
動物では、ポリAリボヌクレアーゼ(PARN)は5'キャップに結合しポリA鎖から塩基を除去する。5'キャップ・ポリA鎖の保護はmRNA分解の制御に重要である。RNAの5'キャップに翻訳開始因子4E(eIF4E)、かつポリA鎖に翻訳開始因子4G(eIF4G)が結合している場合、PARNによる脱アデニル化は減少する。脱アデニル化はRNA結合タンパクにも制御される。一旦ポリA鎖が除去されると5' キャップも除去され、RNAは分解される。酵母からは、脱アデニル化に関わると見られる他の酵素も見つかっている[42]。
代替ポリアデニル化
[編集]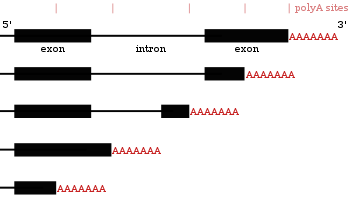
多くのコード遺伝子は複数のポリアデニル化部位を持つため、3' 末端が異なる複数のmRNAをコードしているといえる[24][43][44]。代替ポリA化は3' 非翻訳領域の長さを変更するため、それが含むmiRNA結合部位も変更される[16][45]。転写産物を安定化するmiRNAもあるが、miRNAは翻訳を抑制し、それが結合したmRNAの分解を促進する傾向がある[46][47]。代替ポリA化はコーディング領域を短縮し、別のタンパクを作ることもあるが[48][49]、3' 非翻訳領域の短縮よりは珍しい[24]。
ポリA化部位の選択は細胞外からの刺激に影響される他、ポリA化酵素の発現にも依存する[50][51]。例えば、マクロファージのリポ多糖(免疫応答を起こす細菌分泌物)への反応として、切断刺激因子(CstF)のサブユニットCstF-64の発現が上昇する。この結果ポリA化部位が変更され、防御タンパクmRNA(例えばリゾチームやTNF-α)の3' 非翻訳領域の調節エレメントが除去される。このようなmRNAは長い半減期を持ち、より多くのタンパクを作る[50]。ポリA化機構以外のRNA結合タンパクもポリA化部位の選択に影響し[52][53][54][55]、例えばポリA化シグナルの近くをDNAメチル化する、などの方法で行われる[56]。
細胞質ポリアデニル化
[編集]動物の初期胚発生、または神経細胞のシナプス後段では細胞質でのポリA化が行われる。短縮したポリA鎖を伸長することで、mRNAを翻訳することができる[38][57]。短縮した鎖は約20塩基だが、伸長された鎖は80–150塩基になる[3]。
マウス初期胚では母親由来のRNAを細胞質ポリA化することで、2細胞期の途中(ヒトでは4細胞期)まで転写が始まらないにもかかわらず、細胞は成長することができる[58][59]脳では、細胞質ポリA化は記憶の形成に重要な長期増強に関わり、学習中に活性化される[3][60]。
細胞質ポリA化はRNA結合タンパクCPSF・CPEBを必要とし、Pumilioのような他のRNA結合タンパクも関わる[61]。細胞の種類により、核と同じポリAポリメラーゼ(PAP)・細胞質ポリメラーゼのGLD-2のどちらかが用いられる[62]。
真核生物のRNA分解
[編集]少なくとも酵母では、tRNA・rRNA・snRNA・snoRNAを含む多くの非コードRNAで、ポリA化はRNA分解を促進する[63]。核においてはTRAMP複合体により、約40個の塩基が3' 末端に付加されることでポリA化が行われ[64]、その後エキソソーム複合体により分解される[65]。ポリA鎖はヒトrRNA断片からも発見され、Aのみのホモポリマー・ほぼAのヘテロポリマーの両方が見られる[66]。
原核生物、細胞小器官
[編集]多くの細菌はmRNA・非コードRNA双方をポリA化できる。ポリA鎖はデグラドソームの2つのRNA分解酵素、PNPaseとRNaseEの働きを促進する。特有の二次構造が3' 末端をブロックしていても、ポリA鎖でRNAを伸長することでPNPaseは新しい3' 末端に結合することができる。伸長と分解を繰り返すことで、PNPaseは少しずつ二次構造を解体していく。ポリA鎖はエンドリボヌクレアーゼでの分解も促進する[67]。細菌のポリA鎖は約30塩基の長さである[68]。
動物・トリパノソーマのミトコンドリアは安定なポリA鎖・不安定なポリA鎖双方を持つ。不安定な鎖はmRNAと非コードRNA双方で見られる。ポリA鎖は平均43塩基である。安定な鎖では、ゲノムが終止コドン(UAA)のU・UAまでしかコードしていないため、ポリA鎖は終止コドンの一部となっている。植物ミトコンドリアは不安定なポリA鎖しかもたず、酵母ミトコンドリアはポリA鎖を全く持たない[69]。
多くの細菌・ミトコンドリアはポリAポリメラーゼを持つが、PNPase自身もポリA化を行う。この酵素は細菌[70]、ミトコンドリア[71]、葉緑体[72]、古細菌エキソソーム複合体の構成部品に見られる[73]。Aへの選択性は完全ではないが、ほぼAのみを用いて3' 末端を伸長できる。葉緑体でも細菌のように、PNPaseによるポリA化はRNA分解を促進し[74]、おそらく古細菌でも同じである[69]。
進化
[編集]ポリA化はあらゆる生物に見られるわけではない[75][76]。だが、全ドメインの大部分の生物がこの機能を持つことから、全生物の共通祖先がおそらく何らかのポリA化機構を持っていたと推定される[68]。mRNAをポリA化しない生物は、進化の過程でこの機能を喪失したと考えられる。その実例は細菌Mycoplasma gallisepticum ・高度好塩菌Haloferax volcaniiから得られたmRNAのみで、真核生物では確認されていない[77][78]。
最も古いポリA化酵素は、RNAを分解する複合体(細菌ではデグラドソーム、古細菌ではエキソソーム複合体)の一部であるPNPaseである[79]。この酵素はRNAを3' 末端から加リン酸分解し、NDPに変換する。この反応は可逆なため、RNAにヌクレオチドを付加して伸長させることもできる。エネルギー通貨であるATPが他のNTPより高濃度であるため、伸長した鎖はAリッチである。RNA分解へのAリッチ鎖の関与が、その後のポリAポリメラーゼ(純粋なポリA鎖の合成酵素)の進化を促したことが示唆されている[80]。
ポリAポリメラーゼの起源は古くなく、tRNAの3' 末端を終端するCCA付加酵素から、細菌と真核生物で独立に進化した。その活性ドメインは他のポリメラーゼと相同である[65]。真核生物への、細菌のCCA付加酵素遺伝子の水平伝播により、古細菌様CCA付加酵素がポリAポリメラーゼに機能を変更することができたと推測されている[68]。古細菌や藍藻はポリAポリメラーゼを進化させなかった[80]。
正の制御と負の制御
[編集]上で述べられてきたように、最近では、mRNAのポリ(A)尾部の長さを調節することが、転写後の段階で遺伝子発現を制御する効率的な手段であることが明らかになってきた。実際、発生初期では転写は抑制されており、遺伝子発現は主に細胞質のポリアデニル化によって制御されている。 体細胞では、デアデニル化による負の制御のメカニズムの解明がかなり進んでいる。しかし、ポリ(A)尾部の伸長による正の制御については、観察された長さの増加が、新しいmRNAの合成によるものなのか、デアデニル化の減少によるものなのか、それとも細胞質ポリアデニル化によるものなのかの区別が困難なため、あまり研究されていない。このため、デアデニル化酵素が抑制された条件下で転写パルスチェイス解析を行う方法の開発なども行われてきた[81]。
沿革
[編集]ポリA化は1960年、細胞核抽出物内の酵素が、ATPをポリアデニンに変換したことから発見された[82][83]。多様な細胞から見つかっていたにもかかわらず、1971年にポリA配列がmRNAから見つかるまでその機能は不明のままだった[84][85]。当初は3' 末端をヌクレアーゼから保護するだけと考えられていたが、その後に核外移送や翻訳の際に果たす役割が解明された。関与するポリメラーゼは1960年代に精製され、1970年代に特定されたが、それを制御する無数の補助タンパクが発見されたのは1990年代始めになってからだった[84]。
参照
[編集]出典
[編集]- ^ a b Proudfoot, Nick J.; Furger, Andre; Dye, Michael J. (2002). “Integrating mRNA Processing with Transcription”. Cell 108 (4): 501–12. doi:10.1016/S0092-8674(02)00617-7. PMID 11909521.
- ^ a b Guhaniyogi, J; Brewer, G (2001). “Regulation of mRNA stability in mammalian cells”. Gene 265 (1–2): 11–23. doi:10.1016/S0378-1119(01)00350-X. PMID 11255003.
- ^ a b c Richter, Joel D. (1999). “Cytoplasmic Polyadenylation in Development and Beyond”. Microbiology and Molecular Biology Reviews 63 (2): 446–56. PMC 98972. PMID 10357857.
- ^ Steege, Deborah A. (2000). “Emerging features of mRNA decay in bacteria”. RNA 6 (8): 1079–90. doi:10.1017/S1355838200001023. PMC 1369983. PMID 10943888.
- ^ Anderson, James T. (2005). “RNA Turnover: Unexpected Consequences of Being Tailed”. Current Biology 15 (16): R635–8. doi:10.1016/j.cub.2005.08.002. PMID 16111937.
- ^ Stevens, A (1963). “Ribonucleic Acids-Biosynthesis and Degradation”. Annual Review of Biochemistry 32: 15–42. doi:10.1146/annurev.bi.32.070163.000311. PMID 14140701.
- ^ Lehninger, Albert L.; Nelson, David L.; Cox, Michael M., eds (1993). Principles of biochemistry (2nd ed.). New York: Worth. ISBN 978-0-87901-500-8[要ページ番号]
- ^ Abaza, I.; Gebauer, F. (2008). “Trading translation with RNA-binding proteins”. RNA 14 (3): 404–9. doi:10.1261/rna.848208. PMC 2248257. PMID 18212021.
- ^ Mattick, J. S. (2006). “Non-coding RNA”. Human Molecular Genetics 15 (90001): R17–29. doi:10.1093/hmg/ddl046.
- ^ a b Hunt, Arthur G; Xu, Ruqiang; Addepalli, Balasubrahmanyam; Rao, Suryadevara; Forbes, Kevin P; Meeks, Lisa R; Xing, Denghui; Mo, Min et al. (2008). “Arabidopsis mRNA polyadenylation machinery: comprehensive analysis of protein-protein interactions and gene expression profiling”. BMC Genomics 9: 220. doi:10.1186/1471-2164-9-220. PMC 2391170. PMID 18479511.
- ^ a b Davila Lopez, M.; Samuelsson, T. (2007). “Early evolution of histone mRNA 3' end processing”. RNA 14 (1): 1–10. doi:10.1261/rna.782308. PMC 2151031. PMID 17998288.
- ^ Marzluff, William F.; Gongidi, Preetam; Woods, Keith R.; Jin, Jianping; Maltais, Lois J. (2002). “The Human and Mouse Replication-Dependent Histone Genes”. Genomics 80 (5): 487–98. doi:10.1016/S0888-7543(02)96850-3. PMID 12408966.
- ^ Saini, H. K.; Griffiths-Jones, S.; Enright, A. J. (2007). “Genomic analysis of human microRNA transcripts”. Proceedings of the National Academy of Sciences 104 (45): 17719–24. doi:10.1073/pnas.0703890104.
- ^ Yoshikawa, M. (2005). “A pathway for the biogenesis of trans-acting siRNAs in Arabidopsis”. Genes & Development 19 (18): 2164–75. doi:10.1101/gad.1352605. PMC 1221887. PMID 16131612.
- ^ Amaral, Paulo P.; Mattick, John S. (2008). “Noncoding RNA in development”. Mammalian Genome 19 (7–8): 454–92. doi:10.1007/s00335-008-9136-7. PMID 18839252.
- ^ a b Liu, D.; Brockman, J. M.; Dass, B.; Hutchins, L. N.; Singh, P.; McCarrey, J. R.; MacDonald, C. C.; Graber, J. H. (2006). “Systematic variation in mRNA 3'-processing signals during mouse spermatogenesis”. Nucleic Acids Research 35 (1): 234–46. doi:10.1093/nar/gkl919. PMC 1802579. PMID 17158511.
- ^ Lutz, Carol S. (2008). “Alternative Polyadenylation: A Twist on mRNA 3′ End Formation”. ACS Chemical Biology 3 (10): 609–17. doi:10.1021/cb800138w. PMID 18817380.
- ^ a b Beaudoing, E.; Freier, S; Wyatt, JR; Claverie, JM; Gautheret, D (2000). “Patterns of Variant Polyadenylation Signal Usage in Human Genes”. Genome Research 10 (7): 1001–10. doi:10.1101/gr.10.7.1001. PMC 310884. PMID 10899149.
- ^ Brown, Kirk M; Gilmartin, Gregory M (2003). “A Mechanism for the Regulation of Pre-mRNA 3′ Processing by Human Cleavage Factor Im”. Molecular Cell 12 (6): 1467–76. doi:10.1016/S1097-2765(03)00453-2. PMID 14690600.
- ^ Yang, Q.; Gilmartin, G. M.; Doublie, S. (2010). “Structural basis of UGUA recognition by the Nudix protein CFIm25 and implications for a regulatory role in mRNA 3' processing”. Proceedings of the National Academy of Sciences 107 (22): 10062–7. doi:10.1073/pnas.1000848107.
- ^ Yang, Qin; Coseno, Molly; Gilmartin, Gregory M.; Doublié, Sylvie (2011). “Crystal Structure of a Human Cleavage Factor CFIm25/CFIm68/RNA Complex Provides an Insight into Poly(A) Site Recognition and RNA Looping”. Structure 19 (3): 368–77. doi:10.1016/j.str.2010.12.021. PMC 3056899. PMID 21295486.
- ^ Venkataraman, K. (2005). “Analysis of a noncanonical poly(A) site reveals a tripartite mechanism for vertebrate poly(A) site recognition”. Genes & Development 19 (11): 1315–27. doi:10.1101/gad.1298605. PMC 1142555. PMID 15937220.
- ^ a b Millevoi, Stefania; Loulergue, Clarisse; Dettwiler, Sabine; Karaa, Sarah Zeïneb; Keller, Walter; Antoniou, Michael; Vagner, StéPhan (2006). “An interaction between U2AF 65 and CF Im links the splicing and 3′ end processing machineries”. The EMBO Journal 25 (20): 4854–64. doi:10.1038/sj.emboj.7601331. PMC 1618107. PMID 17024186.
- ^ a b c Shen, Y.; Ji, G.; Haas, B. J.; Wu, X.; Zheng, J.; Reese, G. J.; Li, Q. Q. (2008). “Genome level analysis of rice mRNA 3'-end processing signals and alternative polyadenylation”. Nucleic Acids Research 36 (9): 3150–61. doi:10.1093/nar/gkn158. PMC 2396415. PMID 18411206.
- ^ Glover-Cutter, Kira; Kim, Soojin; Espinosa, Joaquin; Bentley, David L (2007). “RNA polymerase II pauses and associates with pre-mRNA processing factors at both ends of genes”. Nature Structural & Molecular Biology 15 (1): 71–8. doi:10.1038/nsmb1352.
- ^ Stumpf, G.; Domdey, H. (1996). “Dependence of Yeast Pre-mRNA 3'-End Processing on CFT1: A Sequence Homolog of the Mammalian AAUAAA Binding Factor”. Science 274 (5292): 1517–20. doi:10.1126/science.274.5292.1517. PMID 8929410.
- ^ Iseli, C.; Stevenson, B. J.; De Souza, S. J.; Samaia, H. B.; Camargo, A. A.; Buetow, K. H.; Strausberg, R. L.; Simpson, A. J.G. et al. (2002). “Long-Range Heterogeneity at the 3' Ends of Human mRNAs”. Genome Research 12 (7): 1068–74. doi:10.1101/gr.62002. PMC 186619. PMID 12097343.
- ^ Balbo, Paul B.; Bohm, Andrew (2007). “Mechanism of Poly(A) Polymerase: Structure of the Enzyme-MgATP-RNA Ternary Complex and Kinetic Analysis”. Structure 15 (9): 1117–31. doi:10.1016/j.str.2007.07.010. PMC 2032019. PMID 17850751.
- ^ Viphakone, N.; Voisinet-Hakil, F.; Minvielle-Sebastia, L. (2008). “Molecular dissection of mRNA poly(A) tail length control in yeast”. Nucleic Acids Research 36 (7): 2418–33. doi:10.1093/nar/gkn080. PMC 2367721. PMID 18304944.
- ^ Wahle, Elmar (1995). "Poly(A) Tail Length Control Is Caused by Termination of Processive Synthesis". Journal of Biological Chemistry. 270 (6): 2800–2808. doi:10.1074/jbc.270.6.2800 (inactive 18 March 2010). PMID 7852352。
- ^ Dichtl, B.; Blank, D; Sadowski, M; Hübner, W; Weiser, S; Keller, W (2002). “Yhh1p/Cft1p directly links poly(A) site recognition and RNA polymerase II transcription termination”. The EMBO Journal 21 (15): 4125–35. doi:10.1093/emboj/cdf390. PMC 126137. PMID 12145212.
- ^ Nag, Anita; Narsinh, Kazim; Martinson, Harold G (2007). “The poly(A)-dependent transcriptional pause is mediated by CPSF acting on the body of the polymerase”. Nature Structural & Molecular Biology 14 (7): 662–9. doi:10.1038/nsmb1253.
- ^ Coller, J. M.; Gray, N. K.; Wickens, M. P. (1998). “mRNA stabilization by poly(A) binding protein is independent of poly(A) and requires translation”. Genes & Development 12 (20): 3226–35. doi:10.1101/gad.12.20.3226.
- ^ a b Siddiqui, N.; Mangus, D. A.; Chang, T.-C.; Palermino, J.-M.; Shyu, A.-B.; Gehring, K. (2007). “Poly(A) Nuclease Interacts with the C-terminal Domain of Polyadenylate-binding Protein Domain from Poly(A)-binding Protein”. Journal of Biological Chemistry 282 (34): 25067–75. doi:10.1074/jbc.M701256200. PMID 17595167.
- ^ Vinciguerra, Patrizia; Stutz, FrançOise (2004). “mRNA export: an assembly line from genes to nuclear pores”. Current Opinion in Cell Biology 16 (3): 285–92. doi:10.1016/j.ceb.2004.03.013. PMID 15145353.
- ^ Gray, N. K.; Coller, JM; Dickson, KS; Wickens, M (2000). “Multiple portions of poly(A)-binding protein stimulate translation in vivo”. The EMBO Journal 19 (17): 4723–33. doi:10.1093/emboj/19.17.4723. PMC 302064. PMID 10970864.
- ^ Meaux, S.; Van Hoof, A (2006). “Yeast transcripts cleaved by an internal ribozyme provide new insight into the role of the cap and poly(A) tail in translation and mRNA decay”. RNA 12 (7): 1323–37. doi:10.1261/rna.46306. PMC 1484436. PMID 16714281.
- ^ a b Meijer, H. A.; Bushell, M.; Hill, K.; Gant, T. W.; Willis, A. E.; Jones, P.; De Moor, C. H. (2007). “A novel method for poly(A) fractionation reveals a large population of mRNAs with a short poly(A) tail in mammalian cells”. Nucleic Acids Research 35 (19): e132–e132. doi:10.1093/nar/gkm830.
- ^ Lehner, B.; Sanderson, CM (2004). “A Protein Interaction Framework for Human mRNA Degradation”. Genome Research 14 (7): 1315–23. doi:10.1101/gr.2122004. PMC 442147. PMID 15231747.
- ^ Wu, L. (2006). “From the Cover: MicroRNAs direct rapid deadenylation of mRNA”. Proceedings of the National Academy of Sciences 103 (11): 4034–9. doi:10.1073/pnas.0510928103.
- ^ Cui, J.; Sackton, K. L.; Horner, V. L.; Kumar, K. E.; Wolfner, M. F. (2008). “Wispy, the Drosophila Homolog of GLD-2, Is Required During Oogenesis and Egg Activation”. Genetics 178 (4): 2017–29. doi:10.1534/genetics.107.084558. PMC 2323793. PMID 18430932.
- ^ Wilusz, Carol J.; Wormington, Michael; Peltz, Stuart W. (2001). “The cap-to-tail guide to mRNA turnover”. Nature Reviews Molecular Cell Biology 2 (4): 237–46. doi:10.1038/35067025. PMID 11283721.
- ^ Tian, B.; Hu, J; Zhang, H; Lutz, CS (2005). “A large-scale analysis of mRNA polyadenylation of human and mouse genes”. Nucleic Acids Research 33 (1): 201–12. doi:10.1093/nar/gki158. PMC 546146. PMID 15647503.
- ^ Danckwardt, Sven; Hentze, Matthias W; Kulozik, Andreas E (2008). “3′ end mRNA processing: molecular mechanisms and implications for health and disease”. The EMBO Journal 27 (3): 482–98. doi:10.1038/sj.emboj.7601932. PMC 2241648. PMID 18256699.
- ^ Sandberg, R.; Neilson, J. R.; Sarma, A.; Sharp, P. A.; Burge, C. B. (2008). “Proliferating Cells Express mRNAs with Shortened 3' Untranslated Regions and Fewer MicroRNA Target Sites”. Science 320 (5883): 1643–7. doi:10.1126/science.1155390. PMC 2587246. PMID 18566288.
- ^ Tili, Esmerina; Michaille, Jean-Jacques; Calin, George Adrian (2008). “Expression and function of micro-RNAs in immune cells during normal or disease state”. International Journal of Medical Sciences 5 (2): 73–9. PMC 2288788. PMID 18392144.
- ^ Ghosh, T.; Soni, K.; Scaria, V.; Halimani, M.; Bhattacharjee, C.; Pillai, B. (2008). “MicroRNA-mediated up-regulation of an alternatively polyadenylated variant of the mouse cytoplasmic -actin gene”. Nucleic Acids Research 36 (19): 6318–32. doi:10.1093/nar/gkn624. PMC 2577349. PMID 18835850.
- ^ Alt, F; Bothwell, AL; Knapp, M; Siden, E; Mather, E; Koshland, M; Baltimore, D (1980). “Synthesis of secreted and membrane-bound immunoglobulin mu heavy chains is directed by mRNAs that differ at their 3′ ends”. Cell 20 (2): 293–301. doi:10.1016/0092-8674(80)90615-7. PMID 6771018.
- ^ Tian, B.; Pan, Z.; Lee, J. Y. (2007). “Widespread mRNA polyadenylation events in introns indicate dynamic interplay between polyadenylation and splicing”. Genome Research 17 (2): 156–65. doi:10.1101/gr.5532707. PMC 1781347. PMID 17210931.
- ^ a b Shell, S. A.; Hesse, C; Morris Jr, SM; Milcarek, C (2005). “Elevated Levels of the 64-kDa Cleavage Stimulatory Factor (CstF-64) in Lipopolysaccharide-stimulated Macrophages Influence Gene Expression and Induce Alternative Poly(A) Site Selection”. Journal of Biological Chemistry 280 (48): 39950–61. doi:10.1074/jbc.M508848200. PMID 16207706.
- ^ Danckwardt, Sven; Gantzert, Anne-Susan; Macher-Goeppinger, Stephan; Probst, Hans Christian; Gentzel, Marc; Wilm, Matthias; Gröne, Hermann-Josef; Schirmacher, Peter et al. (2011). “p38 MAPK Controls Prothrombin Expression by Regulated RNA 3′ End Processing”. Molecular Cell 41 (3): 298–310. doi:10.1016/j.molcel.2010.12.032. PMID 21292162.
- ^ Licatalosi, Donny D.; Mele, Aldo; Fak, John J.; Ule, Jernej; Kayikci, Melis; Chi, Sung Wook; Clark, Tyson A.; Schweitzer, Anthony C. et al. (2008). “HITS-CLIP yields genome-wide insights into brain alternative RNA processing”. Nature 456 (7221): 464–9. doi:10.1038/nature07488. PMC 2597294. PMID 18978773.
- ^ Hall-Pogar, T.; Liang, S.; Hague, L. K.; Lutz, C. S. (2007). “Specific trans-acting proteins interact with auxiliary RNA polyadenylation elements in the COX-2 3'-UTR”. RNA 13 (7): 1103–15. doi:10.1261/rna.577707. PMC 1894925. PMID 17507659.
- ^ Danckwardt, Sven; Kaufmann, Isabelle; Gentzel, Marc; Foerstner, Konrad U; Gantzert, Anne-Susan; Gehring, Niels H; Neu-Yilik, Gabriele; Bork, Peer et al. (2007). “Splicing factors stimulate polyadenylation via USEs at non-canonical 3′ end formation signals”. The EMBO Journal 26 (11): 2658–69. doi:10.1038/sj.emboj.7601699. PMC 1888663. PMID 17464285.
- ^ Danckwardt, Sven; Gantzert, Anne-Susan; Macher-Goeppinger, Stephan; Probst, Hans Christian; Gentzel, Marc; Wilm, Matthias; Gröne, Hermann-Josef; Schirmacher, Peter et al. (2011). “p38 MAPK Controls Prothrombin Expression by Regulated RNA 3′ End Processing”. Molecular Cell 41 (3): 298–310. doi:10.1016/j.molcel.2010.12.032. PMID 21292162.
- ^ Wood, A. J.; Schulz, R.; Woodfine, K.; Koltowska, K.; Beechey, C. V.; Peters, J.; Bourc'his, D.; Oakey, R. J. (2008). “Regulation of alternative polyadenylation by genomic imprinting”. Genes & Development 22 (9): 1141–6. doi:10.1101/gad.473408.
- ^ Jung, M.-Y.; Lorenz, L.; Richter, J. D. (2006). “Translational Control by Neuroguidin, a Eukaryotic Initiation Factor 4E and CPEB Binding Protein”. Molecular and Cellular Biology 26 (11): 4277–87. doi:10.1128/MCB.02470-05. PMC 1489097. PMID 16705177.
- ^ Sakurai, Takayuki; Sato, Masahiro; Kimura, Minoru (2005). “Diverse patterns of poly(A) tail elongation and shortening of murine maternal mRNAs from fully grown oocyte to 2-cell embryo stages”. Biochemical and Biophysical Research Communications 336 (4): 1181–9. doi:10.1016/j.bbrc.2005.08.250. PMID 16169522.
- ^ Taft, R (2008). “Virtues and limitations of the preimplantation mouse embryo as a model system”. Theriogenology 69 (1): 10–6. doi:10.1016/j.theriogenology.2007.09.032. PMC 2239213. PMID 18023855.
- ^ Richter, J (2007). “CPEB: a life in translation”. Trends in Biochemical Sciences 32 (6): 279–85. doi:10.1016/j.tibs.2007.04.004. PMID 17481902.
- ^ Piqué, Maria; López, José Manuel; Foissac, Sylvain; Guigó, Roderic; Méndez, Raúl (2008). “A Combinatorial Code for CPE-Mediated Translational Control”. Cell 132 (3): 434–48. doi:10.1016/j.cell.2007.12.038. PMID 18267074.
- ^ Benoit, P.; Papin, C.; Kwak, J. E.; Wickens, M.; Simonelig, M. (2008). “PAP- and GLD-2-type poly(A) polymerases are required sequentially in cytoplasmic polyadenylation and oogenesis in Drosophila”. Development 135 (11): 1969–79. doi:10.1242/dev.021444. PMID 18434412.
- ^ Reinisch, Karin M; Wolin, Sandra L (2007). “Emerging themes in non-coding RNA quality control”. Current Opinion in Structural Biology 17 (2): 209–14. doi:10.1016/j.sbi.2007.03.012. PMID 17395456.
- ^ Lacava, John; Houseley, Jonathan; Saveanu, Cosmin; Petfalski, Elisabeth; Thompson, Elizabeth; Jacquier, Alain; Tollervey, David (2005). “RNA Degradation by the Exosome Is Promoted by a Nuclear Polyadenylation Complex”. Cell 121 (5): 713–24. doi:10.1016/j.cell.2005.04.029. PMID 15935758.
- ^ a b Martin, G.; Keller, W. (2007). “RNA-specific ribonucleotidyl transferases”. RNA 13 (11): 1834–49. doi:10.1261/rna.652807. PMC 2040100. PMID 17872511.
- ^ Slomovic, S.; Laufer, D; Geiger, D; Schuster, G (2006). “Polyadenylation of ribosomal RNA in human cells”. Nucleic Acids Research 34 (10): 2966–75. doi:10.1093/nar/gkl357. PMC 1474067. PMID 16738135.
- ^ Régnier, Philippe; Arraiano, Cecília Maria (2000). “Degradation of mRNA in bacteria: emergence of ubiquitous features”. BioEssays 22 (3): 235–44. doi:10.1002/(SICI)1521-1878(200003)22:3<235::AID-BIES5>3.0.CO;2-2. PMID 10684583.
- ^ a b c Anantharaman, V.; Koonin, EV; Aravind, L (2002). “Comparative genomics and evolution of proteins involved in RNA metabolism”. Nucleic Acids Research 30 (7): 1427–64. doi:10.1093/nar/30.7.1427. PMC 101826. PMID 11917006.
- ^ a b Slomovic, Shimyn; Portnoy, Victoria; Liveanu, Varda; Schuster, Gadi (2006). “RNA Polyadenylation in Prokaryotes and Organelles; Different Tails Tell Different Tales”. Critical Reviews in Plant Sciences 25: 65–77. doi:10.1080/07352680500391337.
- ^ Chang, S. A.; Cozad, M.; MacKie, G. A.; Jones, G. H. (2007). “Kinetics of Polynucleotide Phosphorylase: Comparison of Enzymes from Streptomyces and Escherichia coli and Effects of Nucleoside Diphosphates”. Journal of Bacteriology 190 (1): 98–106. doi:10.1128/JB.00327-07. PMC 2223728. PMID 17965156.
- ^ Nagaike, T; Suzuki, T; Ueda, T (2008). “Polyadenylation in mammalian mitochondria: Insights from recent studies”. Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms 1779 (4): 266–9. doi:10.1016/j.bbagrm.2008.02.001.
- ^ Walter, M.; Kilian, J; Kudla, J (2002). “PNPase activity determines the efficiency of mRNA 3'-end processing, the degradation of tRNA and the extent of polyadenylation in chloroplasts”. The EMBO Journal 21 (24): 6905–14. doi:10.1093/emboj/cdf686. PMC 139106. PMID 12486011.
- ^ Portnoy, V.; Schuster, G. (2006). “RNA polyadenylation and degradation in different Archaea; roles of the exosome and RNase R”. Nucleic Acids Research 34 (20): 5923–31. doi:10.1093/nar/gkl763. PMC 1635327. PMID 17065466.
- ^ Yehudai-Resheff, S. (2003). “Domain Analysis of the Chloroplast Polynucleotide Phosphorylase Reveals Discrete Functions in RNA Degradation, Polyadenylation, and Sequence Homology with Exosome Proteins”. The Plant Cell Online 15 (9): 2003–19. doi:10.1105/tpc.013326.
- ^ Sarkar, Nilima (1997). “POLYADENYLATION OFmRNA IN PROKARYOTES”. Annual Review of Biochemistry 66: 173–97. doi:10.1146/annurev.biochem.66.1.173. PMID 9242905.
- ^ Slomovic, S; Portnoy, V; Schuster, G (2008). “Chapter 24 Detection and Characterization of Polyadenylated RNA in Eukarya, Bacteria, Archaea, and Organelles”. RNA Turnover in Prokaryotes, Archaea and Organelles. Methods in Enzymology. 447. pp. 501–20. doi:10.1016/S0076-6879(08)02224-6. ISBN 9780123743770.
- ^ Portnoy, Victoria; Evguenieva-Hackenberg, Elena; Klein, Franziska; Walter, Pamela; Lorentzen, Esben; Klug, Gabriele; Schuster, Gadi (2005). “RNA polyadenylation in Archaea: not observed in Haloferax while the exosome polynucleotidylates RNA in Sulfolobus”. EMBO reports 6 (12): 1188–93. doi:10.1038/sj.embor.7400571. PMC 1369208. PMID 16282984.
- ^ Portnoy, Victoria; Schuster, Gadi (2008). “Mycoplasma gallisepticum as the first analyzed bacterium in which RNA is not polyadenylated”. FEMS Microbiology Letters 283 (1): 97–103. doi:10.1111/j.1574-6968.2008.01157.x. PMID 18399989.
- ^ Evguenieva-Hackenberg, Elena; Roppelt, Verena; Finsterseifer, Pamela; Klug, Gabriele (2008). “Rrp4 and Csl4 Are Needed for Efficient Degradation but Not for Polyadenylation of Synthetic and Natural RNA by the Archaeal Exosome†”. Biochemistry 47 (50): 13158–68. doi:10.1021/bi8012214. PMID 19053279.
- ^ a b Slomovic, S; Portnoy, V; Yehudairesheff, S; Bronshtein, E; Schuster, G (2008). “Polynucleotide phosphorylase and the archaeal exosome as poly(A)-polymerases”. Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms 1779 (4): 247–55. doi:10.1016/j.bbagrm.2007.12.004.
- ^ Ryota Yamagishi; Takeshi Tsusaka; Hiroko Mitsunaga; Takaharu Maehata; Shin-ichi Hoshino (2016-04-07). “The STAR protein QKI-7 recruits PAPD4 to regulate post-transcriptional polyadenylation of target mRNAs”. Nucleic Acids Research (Oxford, England: Oxford University Press) Volume 44 (Issue 6): 2475–2490. ISSN 0305-1048 2021年6月15日閲覧。.
- ^ Edmonds, Mary; Abrams, Richard (1960). “Polynucleotide Biosynthesis: Formation of a Sequence of Adenylate Units from Adenosine Triphosphate by an Enzyme from Thymus Nuclei”. The Journal of Biological Chemistry 235 (4): 1142–9. PMID 13819354.
- ^ Colgan, D. F.; Manley, J. L. (1997). “Mechanism and regulation of mRNA polyadenylation”. Genes & Development 11 (21): 2755–66. doi:10.1101/gad.11.21.2755.
- ^ a b Edmonds, M (2002). “A history of poly A sequences: from formation to factors to function”. Progress in Nucleic Acid Research and Molecular Biology Volume 71. Progress in Nucleic Acid Research and Molecular Biology. 71. pp. 285–389. doi:10.1016/S0079-6603(02)71046-5. ISBN 978-0-12-540071-8.
- ^ Edmonds, M. (1971). “Polyadenylic Acid Sequences in the Heterogeneous Nuclear RNA and Rapidly-Labeled Polyribosomal RNA of HeLa cells: Possible Evidence for a Precursor Relationship”. Proceedings of the National Academy of Sciences 68 (6): 1336–40. doi:10.1073/pnas.68.6.1336.
参考文献
[編集]- Danckwardt, Sven; Hentze, Matthias W; Kulozik, Andreas E (2008). “3′ end mRNA processing: molecular mechanisms and implications for health and disease”. The EMBO Journal 27 (3): 482–98. doi:10.1038/sj.emboj.7601932. PMC 2241648. PMID 18256699.
外部リンク
[編集] ウィキメディア・コモンズには、ポリアデニル化に関するカテゴリがあります。
ウィキメディア・コモンズには、ポリアデニル化に関するカテゴリがあります。
| 遺伝学 | |||||||
|---|---|---|---|---|---|---|---|
| 転写 |
| ||||||
| 翻訳 |
| ||||||
| 調節 | |||||||
| 中心的 人物 | |||||||
|
| |||||||
Text is available under the CC BY-SA 4.0 license; additional terms may apply.
Images, videos and audio are available under their respective licenses.
