ホスファターゼ
ホスファターゼ(Phosphatase;EC 3.1.3)とは、リン酸モノエステル加水分解酵素(ホスホモノエステラーゼ)のことであり、リン酸モノエステルまたはポリリン酸化合物を加水分解し、リン酸と、水酸基を持つ化合物とに変換する脱リン酸化酵素である。ホスファターゼが基質の加水分解を行うことから、加水分解酵素に分類される[1]。プロテインキナーゼなどによるリン酸化とホスファターゼによる脱リン酸化が細胞制御や細胞シグナル伝達(英語版)において多くの役割を担っているので、ホスファターゼは多くの生物的機能にとって重要である[2]。ホスファターゼが分子からリン酸基を取り除く一方で、キナーゼはATPから分子へのリン酸基の転位を進める。同時にキナーゼとホスファターゼは細胞制御ネットワークにとって重要な翻訳後修飾を指示する[3]。 ホスファターゼはリン酸分子から電子伝達体にリン酸基を動かす反応を触媒するホスホリラーゼと混同してはならない。ホスファターゼは細胞の機能調節などに深く関っているため、製薬研究においても関心を集めている[4][5]。
なお広義に、リン酸ジエステル加水分解酵素(ホスホジエステラーゼ)を含めることもある。
ホスファターゼは基質特異性の低いタイプと高いタイプに分けられる。前者にはアルカリホスファターゼや酸性ホスファターゼがあり、p-ニトロフェニルリン酸などの発色基質により活性を測定することができる。後者にはグルコース-1-ホスファターゼやタンパク質ホスファターゼなどがある。
生化学

ホスファターゼはリン酸モノエステルを加水分解し、基質からリン酸部分を取り除く反応を触媒する。反応中に水が割り込み、リン酸イオンにOH基が結合して、もう一方の化合物のヒドロキシ基がプロトン化される。結局、反応全体としてはリン酸モノエステルが分解してリン酸とフリーなヒドロキシ基を持つ分子が生成している[4]。
ホスファターゼは非常に特異的に基質の見かけ上異なる部分に結合するリン酸基を脱リン酸化することができる。ホスファターゼが基質を認識するメカニズムや規則である「ホスファターゼコード」の特定は現在進行中であるが、全てのプロテインホスファターゼの比較分析から、9種の真核生物に共通して「ホスファトーム」(phosphatome)というゲノムが存在することがわかっている[6]。研究によって「ドッキング相互作用」が基質との結合に重要な役割を果たしていることが明らかになった[3]。ホスファターゼは基質上の様々なモチーフを認識し、相互作用している。これらのモチーフは活性部位以外とは親和性が低く、結合しにくい。それぞれのドッキング部位での相互作用は弱いが、多くの相互作用が同時に起こるため、累積効果により結合特異性が生まれる[7]。ドッキング相互作用はアロステリック制御にも影響し、触媒活性を制御する[8]。
機能
キナーゼとは対照的に、ホスファターゼは様々な基質を認識し、反応を触媒する。例えばヒトでは、セリン/トレオニン特異的タンパク質キナーゼはセリン/トレオニンホスファターゼより10倍多い[4]。この不均衡はヒトのホスファトームについての知識、つまり細胞や組織など生体内でのホスファターゼの完全な表現が不充分であることが一因である[3]。多くのホスファターゼは未発見であり、発見されているものについても基質がわかっていないものが多い。しかし研究の進んでいるホスファターゼ/ キナーゼの組み合わせの中では、ホスファターゼは相方のキナーゼよりも形・機能の両面で多くの種類があることがわかっている。これはホスファターゼが不安定であることに起因していると考えられている[4]。

プロテインホスファターゼ
プロテインホスファターゼはタンパク質のアミノ酸残基を脱リン酸化する酵素である。プロテインキナーゼがタンパク質のリン酸化において伝達物質になるのに対し、ホスファターゼはリン酸基を取り除く。この反応は、細胞内で再び信号伝達ができるようにするのに非常に重要である。このようなキナーゼとホスファターゼの連携が膨大な数の細胞の制御を司っている[9]。リン酸化、そして脱リン酸化はタンパク質の翻訳後修飾の中でもっとも多い反応であり、全タンパク質の中で最大約30%がリン酸化されていると考えられている[10][11]。
著名なプロテインホスファターゼにはプロテインホスファターゼ2(PP2A)とPP2Bがある。PP2AはDNA複製や代謝、転写や伸長など様々な制御プロセスに関わっている。カルシニューリンとも呼ばれるPP2BはT細胞の分裂に関わっている。このため、免疫系の抑制を目的とする薬の標的となる[9]。
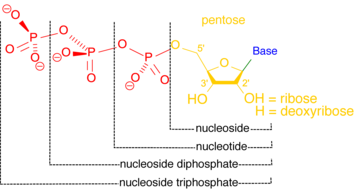
ヌクレオチダーゼ
ヌクレオチダーゼはヌクレオチドを加水分解し、ヌクレオシドとリン酸イオンを生成する反応を触媒する酵素である[12]。ヌクレオチダーゼはヌクレオチドとヌクレオシドのバランスを維持する役割の一部を担っているため、細胞の恒常性を維持するのに重要である[13]。ある種のヌクレオチダーゼは細胞外で、細胞内に運搬してサルベージ経路を通ってヌクレオチドを作るのに使われるヌクレオシドを合成する機能がある[14]。細胞内部ではストレスがかかるとヌクレオチダーゼがエネルギーレベルを維持するのに役立つ。酸素や栄養が供給されなくなった細胞では一次エネルギー源となるATPなどのヌクレオシド三リン酸化物の維持のためより多くのヌクレオチドが異化される [15]。
糖新生でのホスファターゼ
ホスファターゼは炭水化物に対しても糖新生の中間生成物として働く。糖新生はグルコースを炭水化物ではない前駆体から生合成する経路である。多くの生体組織はグルコースからしかエネルギーを得られないので、この経路は重要である[9]。グルコース-6-ホスファターゼとフルクトース-1,6-ビスホスファターゼという2つのホスファターゼが糖新生の不可逆ステップを触媒する[16][17]。それぞれがヘキソースの糖リン酸中間体からリン酸基を取り除く。
分類
酵素委員会はホスファターゼの大分類として104のファミリーを認定している。ホスファターゼは基質特異性と触媒ドメインにおける配列相同性によって分類される[3]。ホスファターゼのファミリーは100以上存在するが、その全てが全体的には同じ加水分解反応を触媒する[1]。
1つのホスファターゼがただ1つの基質にしか反応しないわけではないから、ホスファターゼは現在受け入れられている分類システムとは相容れないところがある。一部のホスファターゼは結合する基質を選ばず、多くの異なる基質を認識し、逆に1つの基質が様々なホスファターゼに認識される[3]。認識する基質のタンパク質によって定義されるプロテインホスファターゼがタンパク質でない基質の脱リン酸化を触媒するケースもある.[4]。同様に、2つの基質に対して特異性を示すプロテインチロシンホスファターゼがチロシン残基だけでなくセリン残基に対しても反応する。したがって、1つのホスファターゼが様々なファミリーの性質を示すことがある[9]。
関連項目
脚注
- ^ a b “ENZYME: 3.1.3.-” (英語). enzyme.expasy.org. 2017年2月21日閲覧。
- ^ Liberti, Susanna; Sacco, Francesca; Calderone, Alberto; Perfetto, Livia; Iannuccelli, Marta; Panni, Simona; Santonico, Elena; Palma, Anita et al. (2013-01-01). “HuPho: the human phosphatase portal” (英語). The FEBS Journal 280 (2): 379–387. doi:10.1111/j.1742-4658.2012.08712.x.
- ^ a b c d e Sacco, Francesca; Perfetto, Livia; Castagnoli, Luisa; Cesareni, Gianni (2012-08-14). “The human phosphatase interactome: An intricate family portrait”. FEBS Letters 586 (17): 2732–2739. doi:10.1016/j.febslet.2012.05.008. PMC 3437441. PMID 22626554.
- ^ a b c d e Li, Xun; Wilmanns, Matthias; Thornton, Janet; Köhn, Maja (2013-05-14). “Elucidating Human Phosphatase-Substrate Networks”. Science Signaling 6 (275): rs10. doi:10.1126/scisignal.2003203.
- ^ Bodenmiller, Bernd; Wanka, Stefanie; Kraft, Claudine; Urban, Jörg; Campbell, David; Pedrioli, Patrick G.; Gerrits, Bertran; Picotti, Paola et al. (2010-12-21). “Phosphoproteomic Analysis Reveals Interconnected System-Wide Responses to Perturbations of Kinases and Phosphatases in Yeast”. Science Signaling 3 (153): rs4. doi:10.1126/scisignal.2001182. PMC 3072779. PMID 21177495.
- ^ Chen, Mark J.; Dixon, Jack E.; Manning, Gerard (2017-04-11). “Genomics and evolution of protein phosphatases” (英語). Science Signaling 10 (474): eaag1796. doi:10.1126/scisignal.aag1796. ISSN 1945-0877. PMID 28400531.
- ^ Roy, Jagoree; Cyert, Martha S. (2009-12-08). “Cracking the Phosphatase Code: Docking Interactions Determine Substrate Specificity”. Science Signaling 2 (100): re9. doi:10.1126/scisignal.2100re9.
- ^ Reményi, Attila; Good, Matthew C; Lim, Wendell A (2006-12-01). “Docking interactions in protein kinase and phosphatase networks”. Current Opinion in Structural Biology. Catalysis and regulation / Proteins 16 (6): 676–685. doi:10.1016/j.sbi.2006.10.008.
- ^ a b c d G., Voet, Judith; W., Pratt, Charlotte (2013-01-01). Fundamentals of biochemistry : life at the molecular level. Wiley. ISBN 9781118129180. OCLC 892195795
- ^ Cohen, Philip (2002-05-01). “The origins of protein phosphorylation”. ネイチャー セルバイオロジー 4 (5): E127–130. doi:10.1038/ncb0502-e127. ISSN 1465-7392. PMID 11988757.
- ^ Tonks, Nicholas K. (2006). “Protein tyrosine phosphatases: from genes, to function, to disease”. ネイチャーレビューズ モルキュラーセルバイオロジー 7 (11): 833–846. doi:10.1038/nrm2039.
- ^ “ENZYME entry 3.1.3.31” (英語). enzyme.expasy.org. 2017年3月21日閲覧。
- ^ Bianchi, V; Pontis, E; Reichard, P (1986). “Interrelations between substrate cycles and de novo synthesis of pyrimidine deoxyribonucleoside triphosphates in 3T6 cells”. 米国科学アカデミー紀要 83: 986–990. doi:10.1073/pnas.83.4.986. PMC 322995. PMID 3456577.
- ^ Zimmermann, Herbert; Zebisch, Matthias; Sträter, Norbert (2012-09-01). “Cellular function and molecular structure of ecto-nucleotidases” (英語). Purinergic Signalling 8 (3): 437–502. doi:10.1007/s11302-012-9309-4. ISSN 1573-9538. PMC 3360096. PMID 22555564.
- ^ Hunsucker, Sally Anne; Mitchell, Beverly S.; Spychala, Jozef (2005-07-01). “The 5'-nucleotidases as regulators of nucleotide and drug metabolism”. Pharmacology & Therapeutics 107 (1): 1–30. doi:10.1016/j.pharmthera.2005.01.003. ISSN 0163-7258. PMID 15963349.
- ^ “ENZYME entry 3.1.3.9” (英語). enzyme.expasy.org. 2017年3月21日閲覧。
- ^ “ENZYME entry 3.1.3.11” (英語). enzyme.expasy.org. 2017年3月21日閲覧。
外部リンク
- Phosphatases - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス
Text is available under the CC BY-SA 4.0 license; additional terms may apply.
Images, videos and audio are available under their respective licenses.
