アクチベーター
アクチベーター(英: activator)は、遺伝子の転写を増加させるタンパク質(転写因子)である[1]。アクチベーターは遺伝子発現を正に制御すると考えられており、遺伝子の転写を促進する機能を持ち、一部の場合では遺伝子の転写が起こるために必要である[1][2][3][4]。アクチベーターの大部分はDNA結合タンパク質であり、エンハンサーまたはプロモーター近位エレメントに結合する[1]。アクチベーターが結合するDNAの部位は、アクチベーター結合部位と呼ばれる[3]。アクチベーターの基本転写装置とのタンパク質間相互作用を行う部分は、活性化ドメイン(activation domain)または活性化領域(activating region)と呼ばれる[1]。
大部分のアクチベーターはプロモーターに近接して存在する調節配列に配列特異的に結合し、基本転写装置(RNAポリメラーゼと基本転写因子)と相互作用することで機能し、基本転写装置のプロモーターへの結合を促進する[1][2][3][4]。他のアクチベーターは、RNAポリメラーゼをプロモーターから解放し、DNAに沿った進行を開始させることで、遺伝子の転写の促進を助ける[2]。また、RNAポリメラーゼはプロモーターから離れた直後に一時停止するが、アクチベーターはこの一時停止したRNAポリメラーゼの転写の継続を可能にすることで機能する場合もある[1][2]。
アクチベーターの活性は調節を受ける。一部のアクチベーターにはアロステリック部位が存在し、特定の分子がこの部位に結合した場合にのみアクチベーターは「オン」状態となって機能する[4]。アクチベーターの翻訳後修飾も活性を調節し、修飾のタイプや修飾されるアクチベーターに依存して活性が増大したり低下したりする[1]。
一部の細胞(多くの場合真核生物)では複数のアクチベーターが結合部位に結合する。これらのアクチベーターは協調的に結合し、相乗的な相互作用を行う[1][2]。
構造
[編集]アクチベータータンパク質は、DNA結合ドメインと活性化ドメインという2つの主要なドメインから構成される。DNA結合ドメインは配列特異的にDNAに結合し、活性化ドメインは他の分子との相互作用によって遺伝子の転写を増加させる機能を持つ[1]。アクチベーターのDNA結合ドメインには、ヘリックスターンヘリックス、ジンクフィンガー、ロイシンジッパーなどさまざまな構造がある。こうしたDNA結合ドメインには特定のDNA配列に対する特異性があり、特定の遺伝子のみを活性化する[1][2][3]。活性化ドメインにもさまざまなタイプがあり、アミノ酸配列によって、アラニンリッチドメイン、グルタミン酸リッチドメイン、酸性ドメインなどに分類される。これらのドメインによる相互作用は特異的ではなく、さまざまな標的分子と相互作用する傾向がある[1]。
アクチベーターにはアロステリック部位が存在する場合もあり、アクチベーター自身の活性化と不活性化を担う[4]。
作用機序
[編集]調節配列への結合
[編集]DNA二重らせんの溝の内部では、塩基対の官能基が露出している。DNAの配列は、水素結合、イオン結合、疎水性相互作用が可能な、独特な表面のパターンを形成する[2]。アクチベーターも特有のアミノ酸配列を持っており、その側鎖がDNAの官能基と相互作用する[2][3]。アクチベータータンパク質を構成するアミノ酸側鎖のパターンは、アクチベーターが結合する特定のDNA配列の表面の特徴と相補的となる[1][2][3]。アクチベータータンパク質のアミノ酸とDNAの官能基との間の相補的な相互作用により、アクチベーターとその調節DNA配列との間にはぴったりとフィットするような特異性が形成される[2]。
アクチベーターの大部分は二重らせんのより広い溝である主溝に結合するが、副溝に結合するものもある[1][2][3]。
アクチベーター結合部位はプロモーターに極めて近接している場合も、遠く離れている場合もある。調節配列が遠く離れている場合、結合したアクチベーターがプロモーターに位置する転写装置と相互作用するために、DNAはループを形成する[2][3]。
原核生物では複数の遺伝子が共に転写されるため(オペロン)、複数の遺伝子が同じ調節配列によって制御されている。真核生物では、遺伝子は個々転写される傾向があり、各遺伝子は自身の調節配列によって制御されている[2]。アクチベーターが結合する調節配列はプロモーターの上流に位置するのが一般的であるが、下流に存在する場合や、真核生物ではイントロン内に存在する場合もある[1][2][3]。
転写を増加させる機能
[編集]調節配列へのアクチベーターの結合は、RNAポリメラーゼの活性を可能にすることで遺伝子の転写を促進する。転写の促進は、転写装置のプロモーターへのリクルートやRNAポリメラーゼの伸長反応の継続の開始など、さまざまな機構で行われる[1][2][3][4]。
リクルート
[編集]アクチベーターによって制御される遺伝子は、必要な転写装置をプロモーター領域へリクルートするためにアクチベーターの調節配列への結合を必要とする[1][2][3]。
アクチベーターとRNAポリメラーゼとの相互作用は、原核生物では大部分が直接的なものであるが、真核生物では間接的なものである。原核生物では、アクチベーターはRNAポリメラーゼのプロモーターへの結合を助けるために直接的な接触を行う傾向がある。真核生物では、アクチベーターは主に他のタンパク質と相互作用し、これらのタンパク質がRNAポリメラーゼと相互作用する[2]。
原核生物
[編集]原核生物において、アクチベーターによって制御される遺伝子のプロモーターはRNAポリメラーゼ単独で強固な結合を行うことはできない。そのため、アクチベータータンパク質がRNAポリメラーゼのプロモーターへの結合を補助する[2][3]。これまさまざまな機構で行われ、アクチベーターは、プロモーターがより露出するようDNAを屈曲させてRNAポリメラーゼが効果的に結合できるようにしたり[3]、RNAポリメラーゼと直接接触してプロモータへ固定したりする[2][3][4]。
真核生物
[編集]真核生物では、アクチベーターは遺伝子の転写を促進するためにさまざまな標的分子をリクルートする。アクチベーターは、他の転写因子や、転写開始に必要なコファクターをリクルートすることができる[1][2]。
アクチベーターはコアクチベーターと呼ばれる分子をリクルートする[1][2]。コアクチベーターは、アクチベーターに代わって、クロマチンの修飾など、転写開始に必要な機能を果たす[1][2]。
真核生物のDNAはより高度に凝縮しているため、アクチベーターはクロマチンを再構成して転写装置がプロモーターにアクセスしやすい状態にするタンパク質をリクルートする傾向がある[1][2]。一部のタンパク質はDNA上のヌクレオソームの配置変えを行い、プロモーター部位を露出させる(ATP依存性クロマチンリモデリング複合体)[1][2]。他のタンパク質は翻訳後修飾によってヒストンとDNAの結合に影響を与え、ヌクレオソームへ強固に巻き付いているDNAを緩める[1][2]。
リクルートされた分子はすべて協調的に機能し、最終的にはRNAポリメラーゼをプロモーター部位にリクルートする[1][2]。
RNAポリメラーゼの解放
[編集]アクチベーターはRNAポリメラーゼに対し、プロモーターを離れてDNA上を移動し、転写開始を行うようシグナル伝達を行うこともできる[2]。RNAポリメラーゼは転写開始直後に一時停止することがあり、この一時停止状態からRNAポリメラーゼを解放するためにアクチベータータンパク質が必要となる[1][2]。この一時停止したRNAポリメラーゼの解放には複数の機構が存在する。アクチベーターは、単にRNAポリメラーゼが移動を継続するようシグナルを伝達することで作用する場合もある[2]。RNAポリメラーゼが転写を継続できないほどDNAが凝縮している際には、DNAを再構築して障壁を除去するタンパク質をアクチベーターがリクルートする場合もある[1][2]。また、RNAポリメラーゼが転写を継続するために必要な転写伸長因子のリクルートを促進する場合もある[1][2]。
アクチベーターの調節
[編集]アクチベーター活性自体もさまざまな方法で調節され、適切な時期とレベルでのアクチベーターによる遺伝子転写促進が保証されている。アクチベーターの活性は、環境刺激や他の細胞内シグナルに応答して増大したり低下したりする[1]。
アクチベータータンパク質の活性化
[編集]アクチベーターは遺伝子の転写を促進する前に「オン」状態になる必要がある場合が多い[2][3][4]。アクチベーターの活性は、DNAの調節部位への結合能力によって制御されている[2][3][4]。アクチベーターのDNA結合ドメインには活性型と不活性型があり、アロステリックエフェクターと呼ばれる分子がアクチベーターのアロステリック部位に結合することで制御される[4]。
アロステリックエフェクターが結合していない状態のアクチベーターは不活性型である。不活性状態ではアクチベーターは特定の調節配列に結合することができず、遺伝子の転写を調節する作用を持たない[4]。
アロステリックエフェクターがアクチベーターのアロステリック部位に結合すると、DNA結合ドメインにコンフォメーション変化が生じ、アクチベーターのDNAへの結合と遺伝子発現の増大が可能となる[2][4]。
翻訳後修飾
[編集]アクチベーターの一部は細胞内での活性に影響する翻訳後修飾が行われる。リン酸化、アセチル化、ユビキチン化などの修飾がアクチベーターの活性を調節することが観察されている。付加される化学基やアクチベーター自身の性質に依存して、翻訳後修飾はアクチベーターの活性を増加させたり低下させたりする。例えば、アセチル化は一部のアクチベーターのDNA結合親和性を増加させることで活性を増大させる。一方、ユビキチン化はアクチベーターがその機能を終えた後の分解の標識となるため、アクチベーターの活性を低下させる[1]。
相乗効果
[編集]原核生物では、1つのアクチベータータンパク質で転写を促進することができる[2][3]。一方真核生物では、通常は複数のアクチベーターが結合部位で組み立てられ、複合体を形成することで転写促進作用を果たす[1][2]。これらのアクチベーターは結合部位に協調的に結合し、あるアクチベーターの結合は他のアクチベーターや転写調節因子の結合部位への親和性を高める。こうした場合、アクチベーターは互いに相乗的な相互作用を行い、複数のアクチベーターが協働することで達成される転写速度は、それらが個々機能する場合の相加効果よりもはるかに高いものとなる[1][2]。
例
[編集]マルトースの異化の調節
[編集]大腸菌Escherichia coliでのマルトースの分解は遺伝子の活性化によって制御されている。マルトースの異化を担う酵素をコードする遺伝子は、アクチベーターの存在下でのみ転写が行われる[3]。
マルトースの分解を担う酵素の転写を制御するアクチベーターは、マルトースの不在下では「オフ」状態となっている[3]。この不活性型の状態では、アクチベーターはDNAに結合して遺伝子の転写を促進することはできない[3][4]。
細胞内にマルトースが存在する場合には、マルトースはアクチベータータンパク質のアロステリック部位に結合し、DNA結合ドメインのコンフォメーション変化を引き起こす。このコンフォメーション変化はアクチベーターを「オン」状態にし、特定の調節配列への結合を可能にする[3][4]。アクチベーターの調節部位への結合はRNAポリメラーゼのプロモーターへの結合を促進して転写を促進し、細胞内に移行したマルトースの分解に必要な酵素の産生を引き起こす[3]。
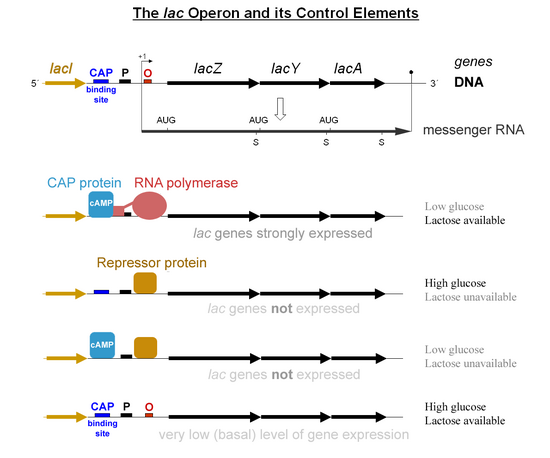
lacオペロンの調節
[編集]カタボライト活性化タンパク質(CAP)またはcAMP受容体タンパク質(CRP)は、大腸菌のlacオペロンの転写を活性化する。環状アデノシン一リン酸(cAMP)はグルコース飢餓状態で産生され、CAPに結合するアロステリックエフェクターとして作用し、CAPのコンフォメーション変化を引き起こしてlacプロモーターに近接する部位への結合を可能にする。その後、CAPはRNAポリメラーゼとの直接的なタンパク質間相互作用によって、RNAポリメラーゼをlacプロモーターにリクルートする[5]。
出典
[編集]- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z aa ab ac ad Ma, Jun (2011). “Transcriptional activators and activation mechanisms”. Protein & Cell 2 (11): 879–888. doi:10.1007/s13238-011-1101-7. ISSN 1674-8018. PMC 4712173. PMID 22180087.
- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z aa ab ac ad ae af ag ah ai aj ak Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Morgan, David; Raff, Martin; Roberts, Keith; Walter, Peter (2015). Molecular Biology of the Cell (Sixth ed.). New York, NY: Garland Science. pp. 373–392. ISBN 978-0-8153-4432-2. OCLC 887605755
- ^ a b c d e f g h i j k l m n o p q r s t u v Madigan, Michael T; Bender, Kelly S; Buckley, Daniel H; Sattley, Matthew W; Stahl, David A (2018). Brock Biology of Microorganisms (Fifteenth ed.). NY, NY: Pearson. pp. 174–179. ISBN 978-0-13-426192-8. OCLC 958205447
- ^ a b c d e f g h i j k l m Griffiths, Anthony J.F.; Gelbart, William M.; Miller, Jeffrey H.; Lewontin, Richard C. (1999). “The Basics of Prokaryotic Transcriptional Regulation” (英語). Modern Genetic Analysis.
- ^ Busby, Steve; Ebright, Richard H (1999-10-22). “Transcription activation by catabolite activator protein (CAP)” (英語). Journal of Molecular Biology 293 (2): 199–213. doi:10.1006/jmbi.1999.3161. ISSN 0022-2836. PMID 10550204.
関連項目
[編集]Text is available under the CC BY-SA 4.0 license; additional terms may apply.
Images, videos and audio are available under their respective licenses.
