ホリデイジャンクション

ホリデイジャンクション(英: Holliday junction)は、4つの核酸配列が塩基対形成によって保持されている核酸構造である。4つのアーム部分のコンフォメーションは、緩衝液の塩濃度と、ジャンクション部位(分岐部位)に最も近い核酸塩基の配列とに依存する。ホリデイジャンクションという名称は、1964年にその存在を提唱した分子生物学者ロビン・ホリデイに由来する。
生物学において、ホリデイジャンクションはさまざまなタイプの遺伝的組換えや二本鎖切断修復において重要な中間体構造として出現する。通常、ジャンクション部分の配列は対称的であり、ジャンクション部位は個々のアームの塩基対が保持されるようなパターンでスライドを行う可能性がある。ホリデイジャンクションに類似した4アームジャンクション構造は一部の機能性RNA分子にもみられる。
自然に存在する可動性ホリデイジャンクションの構造を研究するためのモデルとして、鎖が特定の位置に固定された、非対称的配列からなるホリデイジャンクションが人工的に作り出された。後に、これらのジャンクションはDNAナノテクノロジーにおける基礎的な構造構成要素として利用できることが判明した。複数のホリデイジャンクションを組み合わせることで、高度の構造剛性を持つ形状へ核酸分子をデザインすることができる。
構造
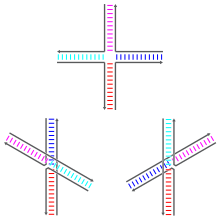
ホリデイジャンクションは、4つの二重らせんアームの同軸的スタッキング(coaxial stacking)のパターンが異なる、さまざまなコンフォメーションで存在する。同軸的スタッキングとは、核酸の露出した塩基間の相互作用によって互いに結合する傾向のことである。ホリデイジャンクションのコンフォメーションには、スタッキングしていないコンフォメーションと2つのスタッキングしたコンフォメーションの3つの可能性がある。スタッキングしていないコンフォメーションはマグネシウムイオン(Mg2+)などの2価カチオンが存在しないときに支配的であり、負に帯電した主鎖間の静電的反発がスタッキングを妨げている。少なくとも 0.1 mMのMg2+の存在下で静電的反発は打ち消され、スタッキング構造が支配的となる。2000年の時点では、マグネシウムなどのカチオンによる静電的遮蔽がジャンクション部位への特異的結合によるものか、溶液中のイオンの多数の散在的結合によるものかは明確には理解されていない[1]。
スタッキングしていないコンフォメーションは、ほぼ平面四角形で、伸長した形状である。一方、スタッキングしたコンフォメーションは2つの連続的な二重らせんドメインからなり、2つのドメインは右巻き方向に約60°回転した関係にある。4つの鎖のうち2つは二重らせんドメインの内部でほぼらせん型を維持したままであり、他の2つは2つのドメイン間を逆平行方向に交差している[1]。
2つのスタッキング型コンフォメーションはどのアームが互いにスタッキングを行うかが異なっており、そのどちらが支配的になるかはジャンクションに最も近接した塩基配列に高度に依存している。一部の配列では2つのコンフォメーション間の平衡状態となるが、他の配列ではどちらかのコンフォメーションが強く好まれる。特に、ジャンクション地点をまたいで A-CCの配列を含むジャンクションは、2つめのシトシンとジャンクション部位のリン酸との間で水素結合が形成されるコンフォメーションを強く好むようである。多くの研究はジャンクションに最も近接した4つの塩基に焦点を当てているが、そこから離れた塩基も明らかにスタッキングしたコンフォメーションに影響を与えうる[1]。
対称的な配列を持つジャンクションでは分岐点は可動的であり、ランダムウォーク過程で移動する。分岐点移動の速度はイオン濃度によって大きく変動し、1ステップの移動にかかる時間は、イオンの非存在下では 0.3−0.4 msであるが、10 mM Mg2+存在下では 270−300 msまで増加する。速度変化は非スタッキング構造に対するスタッキング構造の形成比率と相関している[1]。
ジャンクション地点にニック(一方の鎖の切断)を有するホリデイジャンクションは直交した配向となり、ニックがらせん型の鎖ではなく交差鎖側に位置するスタッキング型コンフォメーションが常に好まれる[1]。
RNAからなるホリデイジャンクションは、高マグネシウム濃度では逆平行方向にスタッキングしたコンフォメーション、中程度の濃度では直交したスタッキングコンフォメーション、低濃度では平行方向にスタッキングしたコンフォメーションになると推定されるが、一方カルシウムイオン存在下では低濃度でも逆平行型コンフォメーションが好まれる[1]。
生物学的機能

ホリデイジャンクションは相同組換えにおける重要な中間体である。相同組換えは、インテグラーゼによる部位特異的組換えと同様、2本の染色体間の遺伝子の移動によって遺伝的多様性を増大させる生物学的過程である。そのほか、二本鎖切断の修復にも関与している[1]。さらにDNA超らせん中の対称配列では、ひずみを緩和するためにホリデイジャンクションを伴う十字型DNA構造が出現することがある[2]。4つのアームからなるジャンクションは、U1 snRNAやタバコ輪点ウイルスのヘアピンリボザイムなどの機能性RNA分子にも存在する。これらは通常二重らせんドメインに対合していないヌクレオチドを含んでいるため、厳密にはホリデイジャンクション構造を取らない[1]。
相同組換えにおいて、ホリデイジャンクションは(ほぼ)同一の配列間で形成され、中央のジャンクションの周囲に対称的な配列が配置される。これによって、ジャンクション部位が移動する分岐点移動の過程が可能となる[1]。ホリデイジャンクションの切断または解消の方法には2通りあり、遺伝子変換が生じているが染色体乗換えは生じていない2つの分子が形成される場合と、乗換えが生じた2つの組換え分子が形成される場合がある。切断の方法に関わらず、ホリデイジャンクションの分岐点移動が起こった領域に関してはすべての産物がヘテロ二本鎖となる[3]。
多くのタンパク質がホリデイジャンクション構造の認識や変形を行う。その1つがホリデイジャンクション解離酵素(Holliday junction resolvase)であり、ジャンクションを(時には配列特異的に)切断する。これらのタンパク質はさまざまな方法でジャンクション構造を変形させるが、多くの場合、スタッキングしていないコンフォメーションへとジャンクションを引っ張ったり、中心部の塩基対を破壊したり、4つのアーム間の角度を変化させたりといったことが行われる。他には、分岐点移動の速度を数桁変化させるタンパク質や、部位特異的組換え酵素などがある[1]。原核生物ではホリデイジャンクション解離酵素はインテグラーゼとヌクレアーゼの2つのファミリーに分類され、それぞれ構造的には類似しているが配列は保存されていない[3]。
真核生物では、相同組換えによってDNAの二本鎖切断を修復する方法には、DSBR経路(double-strand break repair pathway、ダブルホリデイジャンクションモデルとも)とSDSA経路(synthesis-dependent strand annealing pathway)の2つの主要なモデルが存在する[4]。それぞれの過程のアニメーションをこのサイトで見ることができる[5]。
細菌では、DNAの二本鎖切断はRecBCD経路によって修復される。DNA鎖の一方のみに切断が生じている場合はRecF経路によって修復されると考えられている。RecBCD経路もRecF経路も、2つの交差した二本鎖DNA分子の間で一本鎖DNAが交換される分岐点移動の過程と、交差したDNA分子が切り離されて通常の二本鎖状態にもどる解消過程を伴う[6]。細菌では、分岐点移動はRuvABC複合体またはRecGタンパク質によって促進され、これらはATPの加水分解エネルギーを利用してジャンクションの移動を行う分子モーターである。RuvAとRuvBは分岐点移動タンパク質であり、RuvCはジャンクション解離酵素である[1]。
相同組換えはいくつかのウイルスのグループでも生じる。ヘルペスウイルスなどのDNAウイルスでは、組換えは細菌や真核生物と似た切断-再結合(break-and-rejoin)機構で起こる[7]。一部のRNAウイルス、特にレトロウイルスやピコルナウイルス、コロナウイルスといった一本鎖プラス鎖RNAウイルスでは相同組換えの証拠が存在する。インフルエンザウイルスなどの一本鎖マイナス鎖RNAウイルスでも相同組換えが起こるどうかに関しては議論がある[8]。
解消
出芽酵母Saccharomyces cerevisiaeでは、ホリデイジャンクションは4つの異なる経路で解消される。基本的にはin vivoのすべてのホリデイジャンクションがこれらの経路によって解消される[9]。出芽酵母で(そしておそらく哺乳類でも)乗換えの大部分が形成される経路は、EXO1、MLH1-MLH3ヘテロ二量体(MutL-γと呼ばれる)、SGS1(BLMヘリカーゼのオルソログ)が関与する経路である[9]。MLH1-MLH3ヘテロ二量体はホリデイジャンクションに選択的に結合する[10]。この複合体は、超らせんを形成した二本鎖DNAに一本鎖切断を導入するエンドヌクレアーゼである[10][11]。MLH1-MLH3ヘテロ二量体は乗換えを起こした組換え体の形成を促進する[12]。他の3つの経路はそれぞれMUS81-MMS4、SLX1、YEN1が関与するもので、in vivoでのホリデイジャンクションの解消を促進するが、これらの3つのヌクレアーゼ全てが欠けていても乗換えの形成に大きな影響はない。
MLH3(メジャー経路)とMMS4(マイナー経路)の双方を欠失した変異体では、野生型と比較して乗り換えが劇的に(6倍から17倍)低下する。しかし、胞子の生存率は62%とほどほどに高く、染色体分離もおおむね機能しているようである[12]。
出芽酵母、植物、脊椎動物の減数分裂においてはMUS81はマイナー経路の構成要素であるが、テトラヒメナTetrahymena thermophilaではMUS81は支配的ではないにせよ、必須の要素であるようである[13]。MUS81経路は分裂酵母Schizosaccharomyces pombeにおいては支配的経路であるようである[13]。
酵母とヒトでは、MSH4、MSH5タンパク質はヘテロオリゴマー構造(ヘテロ二量体)を形成する[14][15][16]。S. cerevisiaeでは、MSH4とMSH5は減数分裂過程での相同染色体間の乗換えを特異的に促進する[14]。MSH4/MSH5複合体はダブルホリデイジャンクションと呼ばれる2つのホリデイジャンクションからなる構造に結合して安定化し、乗換え型産物が形成される切断パターンを促進する。S. cerevisiaeのMSH4の機能低下型変異体は全ゲノム中の乗換え数を30%減少させ、染色体の乗換えが起こらないパターンの減数分裂を増加させる[17]。この変異体の胞子の生存パターンからは、乗換えが起こっていない染色体でも効率的に分離が起こっていることが示唆される。S. cerevisiaeでは、染色体の正確な分離は相同染色体間の乗換えには完全には依存していないようである。
DNAナノテクノロジーにおける利用

DNAナノテクノロジーは、生細胞内での遺伝情報のキャリアとしてではなく、ナノテクノロジーのための工学的材料として、人工的な核酸構造を設計し作製する技術である。この分野では、より複雑で合理的に設計した構造を作り出すための基礎的構成要素として、分岐したDNA構造が利用される。このようなDNA構造の多くにおいて、ホリデイジャンクションが構成要素となっている。単独のホリデイジャンクション構造では柔軟すぎて大きな規則的アレイへ組み立てることができないため、剛性を持つ「タイル」の作製には複数のホリデイジャンクション構造を持つモチーフが利用される。タイルはその後、より大きな「アレイ」構造へと組み立てられる[19][20]。

そのような構造モチーフで最も一般的なものはダブルクロスオーバー(DX)複合体である。このモチーフには2つのホリデイジャンクションが近接して存在し、より大きなアレイへと自己集合を行い剛性の高い構造となる。DX複合体の構造では、ホリデイジャンクションの二重らせんドメインは最適角度の約60°ではなく、並列するよう強制される。ジャンクションは平行・逆平行のいずれの方向となるようにも設計することができるが、実際的には逆平行型の方が性質が良く、平行型が利用されることは稀である[19][20]。
DX構造モチーフはDNAオリガミにおいても基礎的構造構成要素であり、任意の形状を有する二次元・三次元構造を作るために利用される。DNAオリガミでは、1本の長いスキャフォールド鎖(scaffold、足場)が多数の短いステープル鎖(staple、かすがい)によって目的の形状へと折りたたまれる。組み立てられた際には、スキャフォールド鎖は連続的な二重らせんドメインを形成し、ステープル鎖は交差側の鎖としてホリデイジャンクションに加わる[22]。
一部のタイル構造では、ホリデイジャンクションの自然な60°の角度が維持されている。そのようなアレイの1つとして、平行四辺形型に配置された4つのホリデイジャンクション構造がある。この構造には、原子間力顕微鏡によってジャンクションの角度を可視化することができるという利点が存在する。また、3つのホリデイジャンクションを含む三角形のタイル構造が規則的な三次元アレイを形成するために用いられ、生体分子のX線結晶構造解析に利用される。これらの構造は、張力と圧縮力の双方を利用するテンセグリティの概念に基づいた構造ユニットとの類似性から、テンセグリティ・トライアングル(Tensegrity Triangles)と名付けられている[19][20]。
歴史
ロビン・ホリデイは、トウモロコシ黒穂病菌Ustilago maydisと出芽酵母Saccharomyces cerevisiaeの研究に基づいて、1964年に相同組換えに関するモデルの一部としてジャンクション構造を提唱した。その構造は現在では彼の名前を冠して呼ばれている。ホリデイのモデルは遺伝子変換と染色体乗換えの双方を説明する分子機構をもたらした。ホリデイは提唱した経路が塩基のミスマッチを含むヘテロ二本鎖DNA断片を形成することに気づき、細胞はそのようなミスマッチを修復する機構を持っていると予測した。後に実際にミスマッチ修復機構が発見された[3]。ホリデイのモデル以前に受け入れられていたモデルは"copy-choice"機構と呼ばれるもので、乗換え鎖は部分ごとに異なる鎖から直接合成が行われていると考えられていた[23][24]。
相同組換えに関するホリデイモデルは元々、各親由来のDNAのうちの1本のそれぞれ同じ箇所に一本鎖切断が生じると想定していた。その後、切断された鎖の末端は他方のDNA二重らせんへ向かって移動する。侵入してきた鎖はそこで他の末端と遭遇して連結され、ホリデイジャンクションが形成される。乗換え鎖は当初のパートナー鎖と再アニーリングを行い、元々の相補鎖と置き換わってヘテロ二本鎖断片が形成される。通常、減数分裂の過程では双方のアレルが2コピーずつ生じるが、ミスマッチ修復の際にどちらの鎖が鋳型として利用されるかによって、一方のアレルは3コピー、他方は1コピーとなる可能性がある。これは遺伝子変換として知られる[3]。
ホリデイの最初のモデルは基本的に鎖の連結と交換のみを想定し、双方の染色体のヘテロ二本鎖は相互的な関係となると想定していたが、酵母での実験的データはこれとは矛盾していた。1975年にマシュー・メセルソンとCharley Raddingによって修正されたモデルは、分岐点移動の概念を導入した[23]。1980年代にはさらなる観察によって、二本鎖切断モデル(ジャック・ショスタク、フランクリン・スタールら)や合成依存的単鎖対合モデルなどの代替的機構が提唱された。合成依存的単鎖対合モデルはホリデイジャンクションを伴わないモデルである[3]。
ホリデイジャンクション構造に関する最初の実験的証拠は1970年代後半の電子顕微鏡を用いた研究から得られ、プラスミドとバクテリオファージのDNA像で4アーム構造が明確に可視化された。1980年代には、ホリデイジャンクションの形成開始や結合を担う酵素が同定された。1983年にはネイドリアン・シーマンによって合成オリゴヌクレオチドから人工的なホリデイジャンクション分子が初めて構築され、その物理的性質をより直接的に研究することが可能となった。ホリデイジャンクションに関する初期の分析の多くは、ゲル電気泳動、FRET、ヒドロキシルラジカルやヌクレアーゼによるフットプリンティング研究から得られた。1990年代には、結晶学やNMRを用いた手法や、計算機的な分子モデリングツールが利用されるようになった[1][3][25]。
当初遺伝学者らは、ジャンクションは逆平行型ではなく平行型のコンフォメーションとなると考えていた。このコンフォメーションであれば、相同な二本鎖が互いに近接して配置されるためである[1]。1980年代の化学的解析によってジャンクションは実際には逆平行型のコンフォメーションを好むことが示されたが、その発見には議論の余地があると見なされており、ロビン・ホリデイ自身も当初はその発見を疑っていた[1][3]。In vitroの分子でのX線結晶構造解析のデータによって逆平行型構造は広く受け入れられることとなった。しかしin vivoでの構造に関しては、タンパク質の結合によってジャンクション構造がしばしば変化することもあり、2004年の時点では明らかではない[3]。
DNAナノテクノロジーの概念的基礎は、1980年代初期にネイドリアン・シーマンによって提唱された[26]。当時、DNA複製フォークや可動性ホリデイジャンクションなど、多数の天然の分岐したDNA構造が知られていたが、シーマンは、組み立てられた分子から対称性が除去されるような配列設計によって固定された核酸ジャンクションを形成することが可能であり、原理的にはこのような固定ジャンクションから剛性の高い結晶格子を組み立てることができる、と予見した。この構想を提唱した最初の理論的論文は1982年に発表され、その翌年には固定されたDNAジャンクションの実験的実証がなされた[20][27]。シーマンはより剛性が高く、二次元格子の形成に適したダブルクロスオーバーモチーフを開発し、1998年にエリック・ウィンフリーとともに二次元格子形成を実証した[19]。2006年にポール・ロザムンドは、任意の形状に折り畳まれたDNA構造を容易に安定的に形成する、DNAオリガミ技術を発表し。この手法を用いることで、以前よりもずっと大きな構造を形成することが可能となり、設計や合成の技術的要求はより低いものとなった[28]。ようやく2009年になって、シーマンによって三次元格子の合成が発表された。三次元格子の合成には、彼が取り組み始めてから約30年の歳月を要した[29]。
出典
- ^ a b c d e f g h i j k l m n Lilley, David M. J. (2000). “Structures of helical junctions in nucleic acids”. Quarterly Reviews of Biophysics 33 (2): 109–159. doi:10.1017/S0033583500003590. PMID 11131562.
- ^ Bloomfield, Victor A.; Crothers, Donald M.; Tinoco, Jr., Ignacio (2000). Nucleic acids: structures, properties, and functions. Sausalito, California: University Science Books. p. 468. ISBN 0935702490
- ^ a b c d e f g h “Happy Hollidays: 40th anniversary of the Holliday junction”. Nature Reviews Molecular Cell Biology 5 (11): 937–44. (2004). doi:10.1038/nrm1502. PMID 15520813.
- ^ Sung, P; Klein, H (October 2006). “Mechanism of homologous recombination: mediators and helicases take on regulatory functions”. Nature Reviews Molecular Cell Biology 7 (10): 739–750. doi:10.1038/nrm2008. PMID 16926856.
- ^ “Engelward Laboratory | Animations” (英語). 2020年1月25日閲覧。
- ^ Rocha, EPC; Cornet, E; Michel, B (August 2005). “Comparative and evolutionary analysis of the bacterial homologous recombination systems”. PLoS Genetics 1 (2): e15. doi:10.1371/journal.pgen.0010015. PMC 1193525. PMID 16132081.

- ^ Fleischmann Jr, WR (1996). “Chapter 43”. Medical Microbiology (4th ed.). University of Texas Medical Branch at Galveston. ISBN 0-9631172-1-1
- ^ Boni, MF; de Jong, MD; van Doorn, HR; Holmes, EC; Martin, Darren P. (3 May 2010). Martin, Darren P.. ed. “Guidelines for identifying homologous recombination events in influenza a virus”. PLoS ONE 5 (5): e10434. doi:10.1371/journal.pone.0010434. PMC 2862710. PMID 20454662.

- ^ a b Zakharyevich, K; Tang, S; Ma, Y; Hunter, N (April 2012). “Delineation of joint molecule resolution pathways in meiosis identifies a crossover-specific resolvase”. Cell 149 (2): 334–47. doi:10.1016/j.cell.2012.03.023. PMC 3377385. PMID 22500800.
- ^ a b Ranjha L, Anand R, Cejka P. 2014. The Saccharomyces cerevisiae Mlh1-Mlh3 heterodimer is an endonuclease that preferentially binds to Holliday junctions. J Biol Chem. 2014 Feb 28;289(9):5674-86. doi:10.1074/jbc.M113.533810. PMID 24443562
- ^ “Mlh1-Mlh3, a meiotic crossover and DNA mismatch repair factor, is a Msh2-Msh3-stimulated endonuclease”. J. Biol. Chem. 289 (9): 5664–73. (2014). doi:10.1074/jbc.M113.534644. PMC 3937641. PMID 24403070.
- ^ a b “Genetic analysis of mlh3 mutations reveals interactions between crossover promoting factors during meiosis in baker's yeast”. G3: Genes, Genomes, Genetics 3 (1): 9–22. (2013). doi:10.1534/g3.112.004622. PMC 3538346. PMID 23316435.
- ^ a b “Mus81 nuclease and Sgs1 helicase are essential for meiotic recombination in a protist lacking a synaptonemal complex”. Nucleic Acids Res. 41 (20): 9296–309. (2013). doi:10.1093/nar/gkt703. PMC 3814389. PMID 23935123.
- ^ a b “Conserved properties between functionally distinct MutS homologs in yeast”. J. Biol. Chem. 272 (48): 30345–9. (1997). doi:10.1074/jbc.272.48.30345. PMID 9374523.
- ^ “Cloning and characterization of the human and Caenorhabditis elegans homologs of the Saccharomyces cerevisiae MSH5 gene”. Genomics 53 (1): 69–80. (1998). doi:10.1006/geno.1998.5447. PMID 9787078.
- ^ “hMSH5: a human MutS homologue that forms a novel heterodimer with hMSH4 and is expressed during spermatogenesis”. Cancer Res. 59 (4): 816–22. (1999). PMID 10029069.
- ^ “Variation in crossover frequencies perturb crossover assurance without affecting meiotic chromosome segregation in Saccharomyces cerevisiae”. Genetics 199 (2): 399–412. (2015). doi:10.1534/genetics.114.172320. PMC 4317650. PMID 25467183.
- ^ Mao, Chengde (December 2004). “The emergence of complexity: lessons from DNA”. PLoS Biology 2 (12): 2036–2038. doi:10.1371/journal.pbio.0020431. PMC 535573. PMID 15597116.
- ^ a b c d Seeman, Nadrian C. (June 2004). “Nanotechnology and the double helix”. Scientific American 290 (6): 64–75. doi:10.1038/scientificamerican0604-64. PMID 15195395.
- ^ a b c d Seeman, Nadrian C. (2010). “Nanomaterials based on DNA”. Annual Review of Biochemistry 79: 65–87. doi:10.1146/annurev-biochem-060308-102244. PMC 3454582. PMID 20222824.
- ^ Pan, Keyao; Kim, Do-Nyun; Zhang, Fei; Adendorff, Matthew R.; Yan, Hao; Bathe, Mark (2014-12-03). “Lattice-free prediction of three-dimensional structure of programmed DNA assemblies”. Nature Communications 5: 5578. doi:10.1038/ncomms6578. PMC 4268701. PMID 25470497.
- ^ Saccà, Barbara; Niemeyer, Christof M. (2012). “DNA Origami: The Art of Folding DNA”. Angewandte Chemie International Edition 51 (1): 58–66. doi:10.1002/anie.201105846. PMID 22162047 2015年2月25日閲覧。.
- ^ a b Stahl FW (1 October 1994). “The Holliday junction on its thirtieth anniversary” (PDF). Genetics 138 (2): 241–246. PMC 1206142. PMID 7828807.
- ^ Advances in genetics. Academic Press. (1971). ISBN 9780080568027
- ^ “Caution! DNA Crossing: Crystal Structures of Holliday Junctions”. J Biol Chem 278 (50): 49663–49666. (2003). doi:10.1074/jbc.R300033200. PMID 14563836.
- ^ Pelesko, John A. (2007). Self-assembly: the science of things that put themselves together. New York: Chapman & Hall/CRC. pp. 201, 242, 259. ISBN 978-1-58488-687-7
- ^ Pinheiro, A. V.; Han, D.; Shih, W. M.; Yan, H. (December 2011). “Challenges and opportunities for structural DNA nanotechnology”. Nature Nanotechnology 6 (12): 763–772. doi:10.1038/nnano.2011.187. PMC 3334823. PMID 22056726.
- ^ Rothemund, Paul W. K. (2006). “Scaffolded DNA origami: from generalized multicrossovers to polygonal networks”. In Chen, Junghuei; Jonoska, Natasha; Rozenberg, Grzegorz. Nanotechnology: science and computation. Natural Computing Series. New York: Springer. pp. 3–21. doi:10.1007/3-540-30296-4_1. ISBN 978-3-540-30295-7
- ^ Service, Robert F. (3 June 2011). “DNA nanotechnology grows up”. Science 332 (6034): 1140–1143. doi:10.1126/science.332.6034.1140. PMID 21636754.
外部リンク
Text is available under the CC BY-SA 4.0 license; additional terms may apply.
Images, videos and audio are available under their respective licenses.


